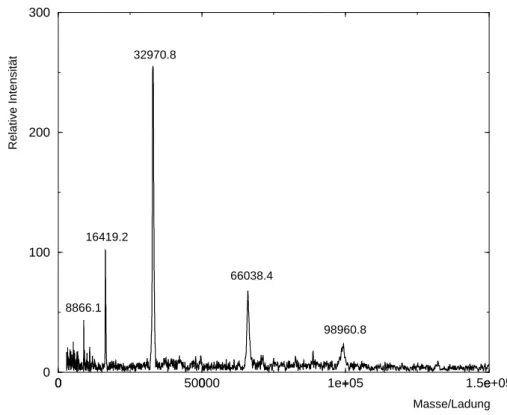
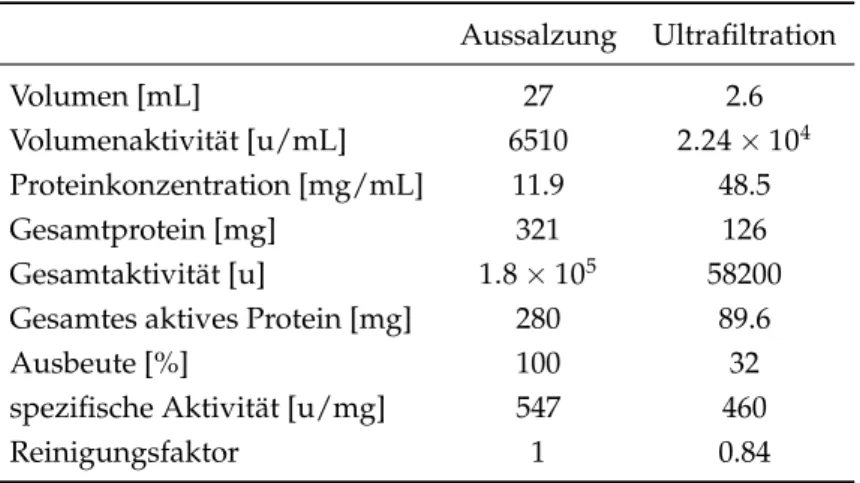
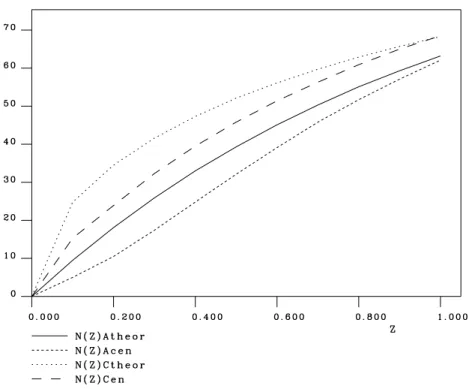
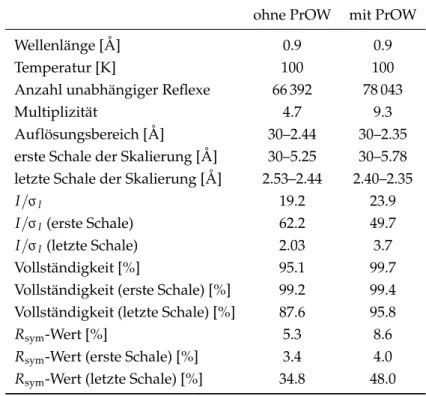
Die Substratbindung der
Volltext
Abbildung

ÄHNLICHE DOKUMENTE
nung verwandt zu sein, insofern sie als eine regelmäßige, aber nicht parallele Verwaehsung zweier gleichartigen Individuen auf- gefasst werden
sätze an, welche aus einer Mischung beider Substanzen bestehen, Erfolgt die Umwandlung der labilen Silbernitratkrystalle, so ver- mag sie, wie bei dem Krystall links oben
Die aus Lösungen sich bildenden Formen beider Proben sind identisch, sie gehören den] monosymmetrischen System an und lassen sich deuten als Basis, Hemipyramide und Orthopinakoid‚
ten Terpentetrahromiden führten nun zu dem Resultat, dass keiner der drei Körper sich in einen andern durch Temperaturiinderung übert'iihren ließ7 dass also, der Annahme von
Es zeigte sich in der Mischzone der Lösungen von gewöhnlicher Bernsteins äure und der fraglichen Substanz keinerl ei Differenz der auftretend en Krystalle und auch
Materieller Eigentumsschutz vermögensrechtlicher Ansprüche nach Art. Zusatzprotokoll EMRK I... 44 Die Historie des Eigentumsschutzes nach
Wenn dann auch über eine Vertretungsagentur keine Aushilfe gefunden werden kann, sodass die Apotheke geschlossen werden müsste, könnte man sich vorstellen, dass ein Gericht
Da die Mehrheit der untersuchten Parameter für die Hypertrophie bei den p47 phox -defizienten Tieren, im Vergleich zu Wildtyp- Tieren, nach MI vermindert war, liegt