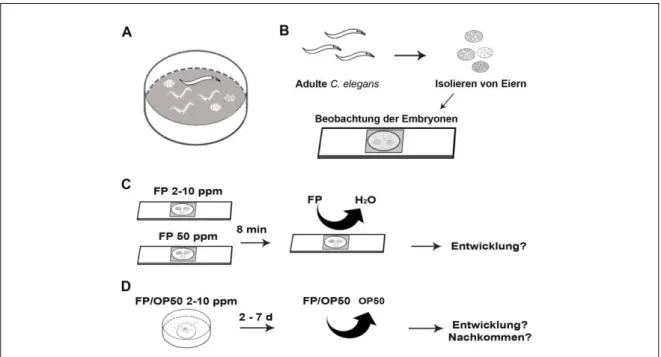
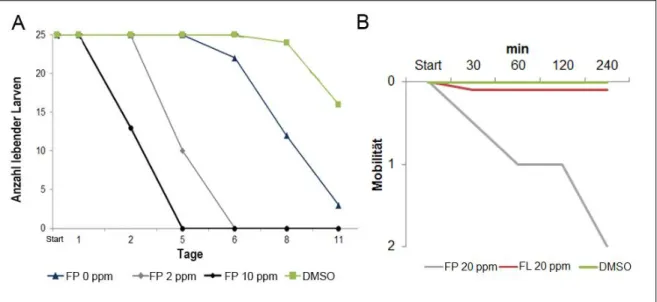
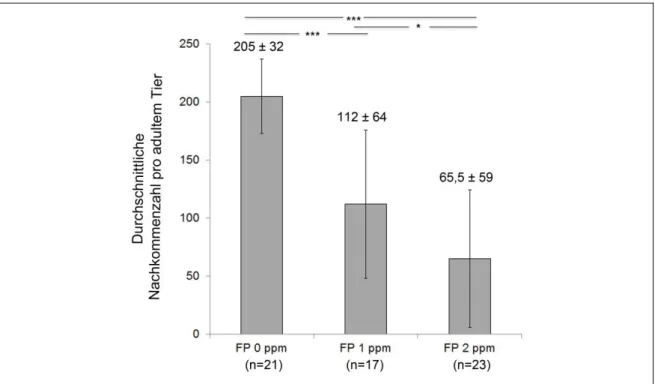
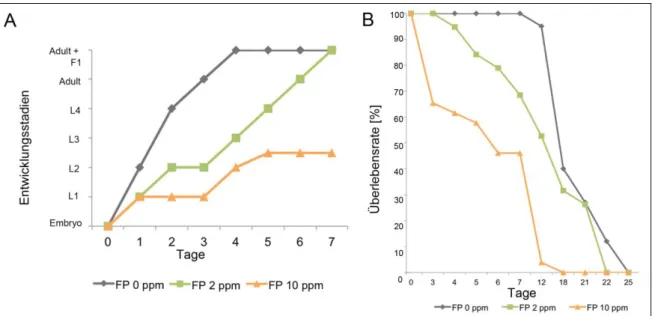
Analyse des Succinat-Dehydrogenase-Inhibitors Fluopyram im Nematoden Caenorhabditis elegans
Volltext
Abbildung




ÄHNLICHE DOKUMENTE
This recapitu- lated the main aspects of tauopathy known from mammalian models of the disease ( 4 , 6 , 13 ), manifested by uncoordinated movement from the first day of
Weiterhin ist auch die geringe mRNA-Menge der Ov-GST3 bei ungestressten Würmern von Bedeutung; während durch semi-quantitative RT-PCR-ELISA Analyse ein extrem starker
zu übertragen (Wood et al. Eine solche Eigenschaft konnte im Rahmen dieser Arbeit, wie schon erwähnt, anhand des rekombinanten Proteins auch für das CePrx1
elegans hingegen die Rolle eines Hungersignals Horvitz et al., 1982, allerdings ist bis dato nichts Weiteres über die Mechanismen bekannt geworden, durch die das Octopamin diese
Dass die Reduktion der DHE-Aufnahme sowohl nach rme-2- als auch vit-6-RNAi nicht drastisch war, ließe sich für die vit-RNAi durch die Redundanz der Vitellogenine
Dabei konnten sowohl Gene identifiziert werden, die durch ver- schiedene Xenobiotika induziert werden als auch welche, die nur durch einen einzelnen Schadstoff in
elegans konnte gezeigt werden, dass die Expression der cyp-Gene durch eine Vielzahl organischer Verbindungen, wie Fluoranthen, Benz(a)Pyren oder PCB52 induziert wird,
Eine Hemmung der Transporterproteine durch Verapamil führte zu einer verstärken Akkumulation des Cadmiums in den Nematoden und macht deren Bedeutung für die zelluläre Entgiftung