zur
Erlangung der Doktorwurde
der
Naturwissenschaftlich-Mat hemati schen
Gesamtfakultat
der
Ruprecht-Karls-Universitat Heidelberg
vorgelegt von
Dipl.-Phys. Kaan Saracoglu
aus Kirkuk
Tag der mundl. Prufung 7. Novemb er2001
Gutachter:
Prof. Dr. Bernd Jahne
Prof. Dr. Dr. Christoph Cremer
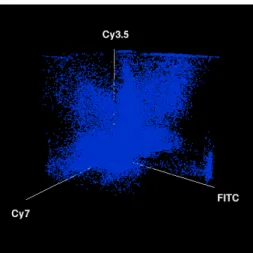
Multiplex-FISHist einevor wenigen Jahren entwickelteMetho de, mit der sich alle
menschlichen Chromosomen mit mehreren Farbstoen kombinatorisch farb en las-
sen [Sp e96a , Sp e96b ,Sch96]. BeiVerwendung von mehralsvier Farb en lassen sich
b ei 31 Kombinationsmoglichkeitenalle Chromosomen in einer eindeutigen kombi-
natorischen Weise farb en und werden mit geeigneten sp ektroskopischen Metho den
anhand ihrer sp ektralen Informationunterscheidbar.
Im Rahmen dieser Arb eit hab e ich ein automatisches Verfahren fur die Analy-
se von Bildern M-FISH markierter Chromosomen und sub chromosomaler Prob en
entwickelt,das signikanteVerb esserungen und robuste Analysen,sowieerweiterte
Moglichkeiten der Analyse mit Hilfe der M-FISH Technologie bietet. Das Verfah-
ren basiert auf Clusteranalyse im Farbraum und kombiniert Farbinformation mit
Ortsinformation,umchromosomaleBereicheimBildvolumenzuidentizieren. Das
Verfahren ist fur die Analyse von Bildern unterschiedlicher M-FISH Exp erimente
-sowohl chromosomal und sub chromosomal, als auch inter- und intrachromosomal-
geeignet. Identizierung einzelner Chromosomen, sowie Volumenund Positionsb e-
stimmungb eidreidimensionalenAufnahmenimInterphase-ZellkernsindimPrinzip
eb enso moglich.
Image Analysis of MFISH - Summary
Multiplex-FISHis a combinatorialstaining technique that allows the simultaneous
detection and discrimination of all human chromosomes. Using at least ve uo-
ro chromes all chromosomes can b e uniquely lab eled in a combinatorial way and
identiedby their sp ecic sp ectral signature.
Within this thesis I develop ed a novel approach for the automated analysis of M-
FISH images, yielding robust classication results and allowing the analysis of M-
FISH images of dierent exp eriments. The metho d combines sp ectral information
with spatial informationto tesselatethe image into regions of similarcolor. Subse-
quentlyaclusteranalysisincolorspace andanal classicationstepare p erformed
to identify the biological targets. This approach is applicable to images of dif-
ferent M-FISH exp eriments, allowing the analysis of interchromosomal as well as
intrachromosomal abnormalities in the genome. It also allows the 3D analysis of
M-FISH lab eled chromosomesininterphase nuclei.
1 Biologische Grundlagen 14
1.1 Die DNA. . . 14
1.2 Gene . . . 16
1.3 Genexpression . . . 17
1.4 Replikationvon DNA-Molekulen. . . 18
1.5 Der Zellzyklus . . . 18
1.6 GenetischeVeranderungen . . . 19
1.7 In Situ Hybridisierung . . . 20
1.8 Multiplex-Fluoreszenzin situ Hybridisierung . . . 21
1.9 ComparativeGenomic Hybridization(CGH) . . . 22
2 Physikalische Grundlagen 24 2.1 Mikroskopie . . . 24
2.1.1 Linsen . . . 24
2.1.2 Auosungsvermogen . . . 25
2.1.3 Axiale Auosung . . . 26
2.1.4 KonfokaleMikrokopie (CLSM). . . 27
2.2 LaserinduzierteFluoreszenz (LIF) . . . 28
2.3 Grenzen der konfokalen Mikroskopie. . . 30
2.3.1 Photonenstatistik . . . 30
2.3.2 Sattigungs- und Bleicheekte . . . 31
2.3.3 Nyquist Theorem . . . 32
2.4 Aufnahmevon M-FISH-Bildern . . . 32
2.4.1 Das MFISH System. . . 32
2.4.2 Das SKY System . . . 34
3 Grundlagen der Bildverarbeitung 37 3.1 Klassizierung . . . 37
3.2 Clusteranalyse . . . 37
3.2.1 K-Means-Algorithmus . . . 39
3.2.2 Fuzzy-Clusteranalyse . . . 39
3.3.1 Einfache Verkettung . . . 42
3.3.2 Hybride Verkettung . . . 42
3.3.3 Zentroide Verkettung . . . 42
3.4 Rauschen . . . 43
3.5 Faltung und Filterung . . . 44
3.5.1 Der Gausslter . . . 45
3.5.2 Der Medianlter . . . 46
3.5.3 Anisotrop er Diusionslter. . . 47
3.6 Dekonvolution . . . 49
4 Methodik 51 4.1 Maximum-Projektion . . . 52
4.2 Hintergrundskorrektur . . . 53
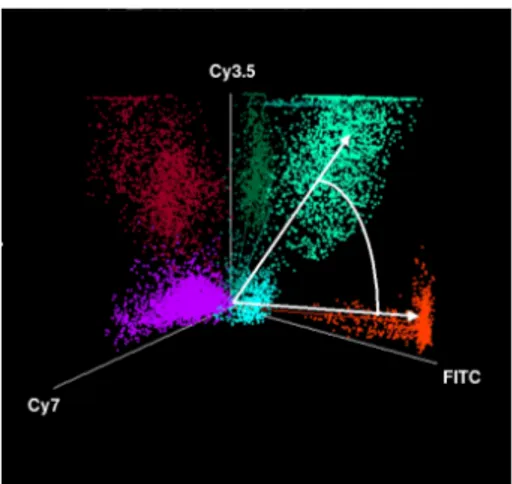
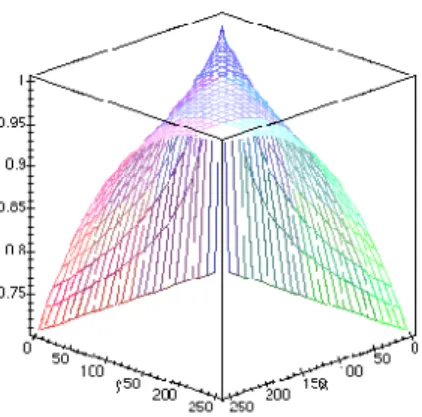
4.3 Farbinformationund Datenraum . . . 55
4.4 Bedeutung der Farb e b ei M-FISH Bildern . . . 56
4.4.1 Winkelals Distanzmass . . . 57
4.4.2 Euklidischer Abstand als Distanzmass . . . 58
4.5 Mo dellbasierteSegmentierung . . . 59
4.5.1 Farbmo dell . . . 60
4.6 Tesselierung . . . 61
4.6.1 Wahl der Startpunkte b eim Regionenwachstum . . . 62
4.7 Clustersuche . . . 63
4.8 Klassizierungder Regionen . . . 64
4.8.1 Merging klassizierterRegionen . . . 66
4.9 Zusammenfassungder Metho dik . . . 66
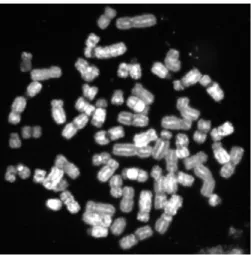
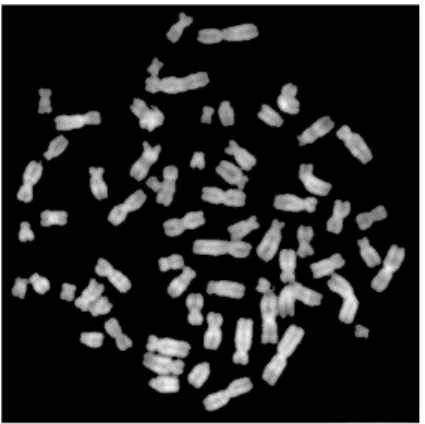
5 Anwendungen 69 5.1 Anwendungen in2D . . . 69
5.1.1 NormaleMetaphase . . . 69
5.1.2 Ab errante Metaphase . . . 70
5.1.3 ChromosomaleBarco des . . . 72
5.1.4 Telomere . . . 73
5.2 Anwendungen in3D . . . 74
5.2.1 3-Farb en Exp eriment mit sieb enChromosomenpaaren in der Interphase . . . 75
6 Ergebnisse und Diskussion 78 6.1 Auosung und Hybridisierungsschema. . . 79
6.2 Klassikationsgute von M-FISHimVergleichmitSKYfurMetapha- sen in 2D . . . 81
6.3.1 Binare Klassikation . . . 82
6.3.2 Klassikation mitEuklidischemAbstand . . . 84
6.3.3 Regionenwachstum mit Euklidischem Abstand als Ahnlich- keitsmass . . . 85
6.3.4 Clustersuche ub erEuklidischenAbstand . . . 87
6.4 ChromosomaleBarco des . . . 89
6.5 Telomere . . . 90
6.6 Anwendungen in3D . . . 91
6.7 ZusammenfassendeDiskussion . . . 95
7 Zusammenfassung 98 8 Bedienung der Software 102 8.1 Die Datei classes . . . 102
8.2 Die Datei menu . . . 104
9 Publikationen im Rahmen dieser Dissertation 109
Robert Frost
"Der Mensch,dieKrone derSchopfung, das klugsteund stolzesteGeschopf,das die
Evolutionhervorgebrachthat,steht,wasseinengenetischenBauplanangeht,fastauf
einerStufemitWurmernund Fliegen."Diesenb emerkenswerteAussagekonnteman
am 12.02.2001 in einer grossen deutschen Tageszeitung lesen. Die Entschlusselung
des menschlichen Genoms brachte zu Tage, dass das menschliche Erbgut oenbar
nur zwischen 26000 und 40000 Gene b esitzt, etwas mehr als der Fadenwurm, der
einen Millimeterlang ist und praktisch
ub er kein Gehirn verfugt. No ch vor einem
Jahr waren Forscher mit einer Zahl von bis zu 130000 Genen ausgegangen. Die-
se Zahl b eruhte auf der Vorstellung, dass ein Gen fur die Synthese eines Proteins
zustandig ist, die Zahl der Gene in etwa derjenigen der Proteine entspricht. Diese
Vorstellung istdamit hinfallig.
DenGenenstehenalsodrei-biszehnmalsovieleProteinegegenub er. Oenbar kann
einGenden BauplanfurverschiedeneProteineliefern. MitdieserErkenntnisgewin-
nen weitere Zweige der Molekularbiologie an Bedeutung: Post- Genomics, das sich
mit der Aktivierung und Deaktivierung von Genen und dem Ableseprozess durch
Botenmolekuleb eschaftigt, und dieProteomik, dieUntersuchung der Proteine, ih-
rer Pro duktion und ihres komplexenWechselspielsin den menschlichenZellen. Die
Erforschung von Proteinen ist jedo ch sehr viel komplexer, da ihre Wirkung nicht
nurauf ihrermolekularenStruktur,sondern auchaufihrerDynamik,ihremZusam-
menspiel b eruht. Eiweisse werden in der Zelle synthetisiert, mo diziert,angehauft
und transp ortiert,sieb eeinussenandereProteineeb enso wieGene. DieAnnahme,
dass Gene und Proteine sich in geradlinigen kausalen Wirkungsketten verhalten,
dass zum Beispiel Gen A fur die Synthese von Protein A sorgt, das dann Protein
B aktiviert, welches wiederum Eiweiss C anschaltet, ist nach dem heutigen Stand
der Erkenntnis
ub erholt. Ihr Verhaltenistviel zufalliger und unstrukturierter. Der
Biologie fehlt jedo ch der theoretische Rahmen, um solcheSysteme zu b eschreib en,
wahrend in der Mathematik und Physik schon zahreicheMetho den existieren, um
solchesVerhalten zuerfassen.
Do ch nichtnurdie geringeZahl der Gene isterstaunlich,sondern auchihre Vertei-
lunginnerhalbdesGenoms. DagibtesausgedehnteBereiche,indenensichgarkeine
Gene b enden, wahrend sieinanderen Abschnittendichtan dichtliegen. Auchdie
Verteilung der Gene auf die Chromosomen ist nicht gleichmassig. Chromosom 18
b eispielsweise ist b esonders genarm und b esteht ub er die Halfte aus solchen Leer-
stellen.
Der Mithilfe von Bioinformatikern ist es zu verdanken, dass man heute ca. 10000
menschlichen Genen eine Funktion zuordnen kann. Etwa 1100 menschliche Gene
sind dafur b ekannt,eineKrankheitauslosen zukonnen, wenn siemutieren,also ihr
genetischerCo deverandertwird. Glucklicherweisegibt eszwarmehralseinhundert
Gene, dieRepaparaturwerkzeuge b ereitstellen,deren Aufgab ees ist,solcheDefekte
bosartige Tumorezuruckzufuhren. Mehr
ub erdieUrsachender Krebsentstehungzu
erfahren und entsprechendeTherapienzu entwickeln,istdahereineder wichtigsten
Aufgab en der medizinischenForschung.
Die b eiKrankheiten auftretenden genetischen Anomaliensind dab ei vielfaltig. Ge-
ne konnen
ub erreprasentiert sein o der ganz fehlen, ab er auch ganze Chromosomen
konneninkrankenZellenfehleno der inmehralszweiKopienauftreten. Sehrhaug
kommtes auch zu inter- und intrachromosomalenUmbauten: Ein DNA Abschnitt
von ChromosomA ndetsich
"
plotzlich\ aufChromosom Bwieder,o der aufeinem
versetztenBereichdesselb en Chromosoms.
Dab ei gibt es verschiedene exp erimentelle Metho den, um die genomische Struk-
tur und Aktivitat aufunterschiedlichergenomischerAuosung sichtbarzumachen.
Allen imfolgenden aufgefuhrtenMetho den liegtdie Technikder Hybridisierung zu-
grunde. Man machtsich hierdie Eigenschaft der DNA-Bausteine (Nukleinsauren)
bzw. Sequenzenzunutze,mitinihrerBasenabfolgekomplementarenSequenzenBin-
dungen einzugehen: Interessierende Bereiche auf den DNA Einzelstrangen konnen
sichtbar gemachtwerden,in demder diesemBereichkomplementareAbschnitt mit
radioaktiveno der uoreszierenden Molekulen markiertwird.
DazuzahlenunteranderemdieComparativeGenomische Hybridisierung(CGH)zur
Analyse der Kopienanzahl chromosomalerRegionen mit einer Auosung von etwa
10Mbp unddieSerielle Analyse von Genexpression zur Untersuchung derAktivitat
einzelnerGene,mithohercytogenetischerAuosungimBereichvoneinigentausend
Basenpaaren. SiendenihreAnwendungimBereichderBio-Chip-Technologieunter
anderem b ei den sogenannten DNA-Microarrays. Ein DNA-Microarray o der DNA-
Chip b esteht aus einem Trager mit einer meist glasernen Ob erache, auf den ra-
sterformig unterschiedlicheNukleinsaurenals Hybridisierungszieleaufgetragen und
xiert werden. Anschliessend gibt man in Losung b endliche komplementare Nu-
kleinsaure-Molekule hinzu. Dab ei handelt es sich in der Regel um die Bausteine
zweierunterschiedlicherDNAs,diezuvormitunterschiedlichenFarbstoenmarkiert
wurden,wob eieinealsReferenzdient. ZweiwichtigeAnwendungendieserTechnolo-
giesinddieUntersuchunggenomischerImbalanzenin der Tumorgenesemitgenomi-
scherDNA,unddieAnalyseder GenexpressionmitmRNA(messengerRib oNucleic
Acid),das Aufschlussgibt
ub erStowechselwege,ReaktionenaufArzneimittelund
Umweltb edingungen. Parallele Abfrage einer grossen Anzahl von Zielstellen in der
DNA, Automatisierungder Chippro duktion,Automatisierungder Auswertung und
die VerizierbarkeiteinzelnerDaten mitherkommlichenmolekularbiologischen Me-
tho den sind dab ei wichtige okonomischeVorzuge der Chip-Technologie.
Eineexp erimentellerAnsatz mitniedrigerer genomischerAuosungimBereichvon
einigenMillionenBasenpaaren istdiesogenannteFluoreszenzin situHybridisierung
zischerDNA-Prob en. EineWeiterentwicklunghiervonistdiesogenannteMultiplex-
Fluoreszenz in situ Hybridisierung (M-FISH),eineTechnikdiees erlaubt,mitHilfe
mehrerer Farbstoedas gesamteGenom -also alle24 Chromosomen-,in einem Ex-
p erimentmit unterschiedlichen Farb en und Farbkombinationenzu markieren. Mit
geeigneten sp ektralen Techniken wird die Identizierung aller Chromosomen einer
Zelle eines Patienten und damit ihre Sortierung in ein sogenanntes Karyogramm
moglich(Karyotypisierung), das Aufschluss ub er die Kopienanzahl der Chromoso-
menund dieinterchromosomalenAb errationengibt. Ab er auchintrachromosomale
Ab errationenundAnomalieninsp ezischenBereicheninnerhalbder Chromosomen
konnenmitdieser Technologie sp ezischgefarbt und untersucht werden.
MitsolchenAnalysenkonnenZusammenhangezwischenKrankheitsbild(Phanotyp)
undgenomischerStruktur(Genotyp)erstelltwerden. FruhdiagnosenundPrognosen
zum Krankheitsverlaufwerden auf der Basis dieser Informationen moglich. Recht-
zeitigetherap eutischeMassnahmenkonnenzumTeilschonvorAusbruchderKrank-
heit eingeleitet werden, was die Chancen einer erfolgreichen Behandlung drastisch
erhoht. AnomalieninderDNAkonnenaufunterschiedlichengenomischenAuosun-
gen auftreten. Es kann daher sein, dass mehrere Exp erimentenotwendig sind, um
alle b ei einer Krankheit sp ezisch auftretenden genetischen Ab errationen zu erfas-
sen. Gewonnene exp erimentelle Daten mussen daher in Datenbanken gesp eichert,
aktualisiertund mitneuen exp erimentellenResultatenerweitert undsynchronisiert
werden. Mit Verfahren aus dem Bereichder kunstlichen Intelligenzlassen sich aus
solchen Datenbanken Strukturen detektieren,die Ruckschlusse auf funktionale Zu-
sammenhangeliefern.
MitfortschreitenderEntwicklunginderBiotechnologie,derzunehmendenDatenut
und Komplexitatder zub ewaltigenden Aufgab enistinden letztenJahrenauchder
Bedarf an automatischen Analyseverfahren zur Fruherkennunggenetischb edingter
Krankheiten rapide angestiegen. Damit erhebt sich die Notwendigkeit interdiszi-
plinarerForschungund Entwicklung-diese Arb eitisteinSchrittindiese Richtung.
Kern dieser Dissertation war die Entwicklung einer robusten, exiblen, automa-
tischen Analysemetho de von M-FISH Bildern. Es sollte nicht nur die zuverlassi-
ge Karyotypisierung von normalen und ab erranten Zellen, sondern auch die Ana-
lyse von alternativen, auf M-FISH basierenden Exp erimenten mit kleinen DNA-
sp ezischen Prob en und Exp erimenten zur Untersuchung von intrachromosomalen
Anomalien erlaub en. Eine Analyse von dreidimensionalen Bildaufnahmen von M-
FISH markierten Chromosomen im Interphase-Zellkern ist eb enso moglich. Mor-
phologische Grossen der einzelnen Chromosomen und ihre relativen Positionen zu-
einander konnen b estimmt werden. Wahrend im zweidimensionalen das Resultat
direktveriziertwerdenkann, istdiesimdreidimensionalenFallschwieriger,da das
Signal-Rausch-VerhaltnishierschlechterunddieFarbinformationdamitdiuser ist.
WeiterhinfuhrenUb erlappungenzwischenChromosomeninnerhalb desZellkernszu
irrefuhrenden Farbsignalen, die zu erkennen nur schwer wenn nicht gar unmoglich
ist. Und nicht zuletzt die dreidimensionaleStruktur macht eine Verizierung
ub er
alle Bereicheaufwendig.
ZentraleBedeutungb eidieserArb eit hattedieAnalyseimmehrdimensionalenFar-
braum unddie Untersuchung der Farbraumcharakteristikb eiunterschiedlichenEx-
p erimenten. Dab ei ging esnichtnur umdieFrage einer robusten Identikation aus
bildverarb eitungstechnischer Sicht, sondern auch um Fragen der exp erimentellen
Moglichkeiten, die farblicheUnterscheidung der Chromosomen und damit die
"
ex-
p erimentelle\Auosungzuoptimieren,umeineoptimaleIdentifkationzuerreichen.
Wichtigab er war esauch,Grenzendieser Technikund Fehlerquellender akkuraten
Identikation zu erkennen und aufzuzeigen. Zu Beginn dieser Arb eit handelte es
sichb eiMFISHumeinesehrneue Technologie. Auffolgende Arb eitenkonntediese
Dissertation aufsetzen:[Sp e96a , Sch96 , Gar96 , Eil98].
BeiHybridisierungsexp erimenten treten schwerquantizierbare Rauschquellen auf.
Das ideale Hybridisierungsexp eriment sollte eine homogene Farbung der biologi-
schen Praparate gewahrleisten. Das ist jedo ch nur selten der Fall, meistens sind
die Intensitatsschwankungen innerhalb kleinerBereichesehr stark. Die Grunde fur
diese Inhomogenitaten sind im Detail no ch nicht genauverstanden. Bei Mehrfach-
hybridisierungen,wieesb eiM-FISHExp erimentenderFallist,kommenno chweite-
re Schwierigkeiten hinzu. BestimmteFluoreszenzkombinationenfuhren b ei einigen
ChromosomenzusehrschwachenHybridisierungen. DieHybridisierungsqualitatder
unterschiedlichenFarbstoeunddieBildaufnahmederunterschiedlichenFarbkanale
mussenaufeinander abgestimmtwerden. Standardisierte Farb eprotokolle und Pro-
b enkitshab ensicherstkurzlichetabliert,weshalbjedesLab or,dasaufdiesemGebiet
arb eitet, zunachst sein eigenes Hybridisierungsprotokoll und -Schema entwickelte.
Das wiederum stellte hohe Anspruche an die Bildverarb eitung und erforderte e-
xible Analysemetho den, um der unterschiedlichen Qualitat der anfallenden Daten
gerechtzuwerden.
Daessichb eiderAnalysegenomischerImbalanzenmitHilfederCGHgleichfallsum
ein Mehruoreszenzexp erimenthandelt,konnten wesentlicheTeileder fur die Ana-
lyse von M-FISH Bildern entwickelten Metho den auch auf die Analyse von DNA-
Microarraysangewendetwerden. DazuwurdeeigenseineSoftwareimRahmeneiner
Diplomarb eit entwickelt[Rud00]. Obwohl der Schwerpunkt dieser Arb eit nichtauf
dieser Entwicklungb eruht,werdenauchfurdiese Anwendung diefurdas Verstand-
nis notwendigenbiologischenund exp erimentellenGrundlagen dargelegt.
Zunachst lege ich die fur diese Arb eit relevanten biologischen und fur die Bildauf-
nahme wichtigen physikalischen Grundlagen in Kapitel 1 und 2 dar. Dazu zahlen
vor allem die Technik der Multiplex - Fluoreszenz in situ Hybridisierung und die
sp ektrale Bildaufnahme am Mikroskop. Anschliessend geb e ich im dritten Kapitel
eineEinfuhrunginrelevantegrundlegendeBildverarb eitungsop erationen. InKapitel
4 b eschreib eichausgehend von der Analysedes Farbraums dievon mirentwickelte
Metho dik, die sich zusammengefasst am Ende des Kapitels wiederndet. Anwen-
dungen, die ich in Kapitel 5 prasentiere, sind im vierten Kapitel zum Teil schon
vorweggenommen, um den Weg der Entwicklung anschaulich und nachvollziehbar
zu dokumentieren. Ergebnisse meiner Metho dik diskutiere ich in Kapitel 6. Eine
etwas ausfuhrlichere Zusammenfassung ndet sich am Ende dieser Arb eit und ist
bilingualgehalten.
Biologische Grundlagen
DieZelleistdiestrukturelleBasisfurjedenOrganismusundweistStowechselsowie
die Fahigkeit,sich zurepro duzierenauf. Man unterscheidetzwischenProkaryonten
und Eukaryonten, letztere weisen eine Zellkern auf und b estehen im wesentlichen
aus dem Nukleus (Zellkern), dem Cytoplasma und der Zellmembran. Im Zellkern
b endet sichdas Chromatin, dieGesamtheit der Chromosomen, und als kompakte
Struktur das Nukleolus (Kernkorp erchen), indem die ribosomale RNA (rRNA) sy-
thetisiert wird. Das Zellkernplasma (Karyoplasma) wird durch die Kernmembran
vom Cytoplasma getrennt. In den Zellen aller Leb ewesen istdie
"
Erbgutinformati-
on\ auf demselb en Makromolekul-der DNA- gesp eichert. Durch Untersuchungen
an Bakterien und ihren Viren 1
ist in den vierziger Jahren erstmals der eindeuti-
ge Nachweisfurdie Funktion der DNA als Trager dieser Informationen, der Gene,
gelungen.
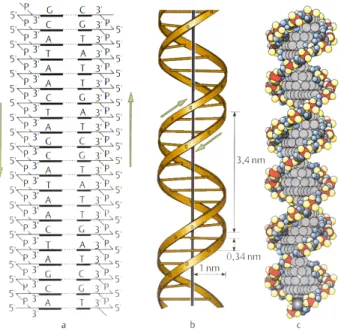
1.1 Die DNA
Die Desoxyrib onukleinsaureisteinlangesPolymermolekul,dasaus kettenartigmit-
einanderverbundenenPurinbasenAdenin,Thymin,GuaninundCytosin,sowiewei-
teren Proteinen, dieals Geruststoe dienen,aufgebaut ist. Der Grundbaustein der
DNA istdas Nukleotid, das sich aus dreiEinheitenzusammensetzt:
einemZucker,der Desoxyrib ose
einer stickstohaltigen Purinbase
und einer Phosphorsaure.
Im Falle der DNA ist der Zucker eine Desoxyribose und die moglichen Basen sind
Adenin (A), Guanin (G), Cytosin(C) und Thymin (T). Bei der RNA dagegen ist
1
Dasentscheidende Exp erimentdazustammtvonO.AveryundMitarb eitern.
funfzigerJahrenhat E.Chargafestgestellt,dass dasVerhaltnisvonA zuT(U)und
das von G zu C immergleichist, weshalb die prozentuale Basenzusammensetzung
jeder DNAvorausgesagt werden kann.
In der Folgezeit zeigten Rontgenstruktur-Analysen, dass die DNA eine regelmassi-
ge Perio dik aufweist und aus zwei Einzelstrangen aufgebaut ist. 1953 schlossen
J.D.Watson und F.Crickauf dieDopp elhelixstruktur.
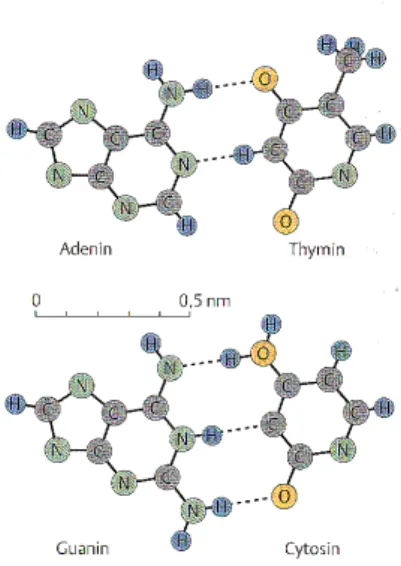
In Abb. 1.2 b) ist die Dopp elhelix als Dopp elspirale gezeigt, deren Einzelstrange
ub erStufen(Wasserstobruckenbindungen)miteinanderverbundensind. DieBander
enthaltendieZucker-Phosphat-Teile,wahrenddieStufen durch dieBasenb eschrie-
b en werden. Allerdingssind nurganz sp ezielleBasenpaarungen, namlichA-T bzw.
G-C, moglich.
Abb.1.1: DieBasen derDNAund ihre Paarung (Quelle:Kni95)
FolglichsinddiezweiBasenstrangekomplementarzueinander. Aufgrunddessenkann
von der Nukleotid-Sequenzdes einen Strangs die des anderen abgeleitetwerden.
Beim Menschen ist die gesamte Erbgut auf 23 Chromosomenpaaren gesp eichert.
Man unterscheidet dab ei zwischen den 44 Autosomen und den zwei Gonosomen,
den Geschlechtschromosomen. DerUnterschiedzwischenMann undFrau b estehtin
genaueinemChromsom: dieFrauhat zweiX-Chromsomen,wahrendder mannliche
Karyotyp ein X- und ein Y-Chromosom aufweist. Menschliche Chromsomen ent-
halten im Mittel 310 9
Basenpaare. Das entspricht einemDNA-Strang mit einer
Lange von 5-10 cm.
c sog.Kalottenmo dell-Darstellung(blau: H,gelb: O, grau: CinderPhospho diesterkette,
hellgrau: Cbzw.N in den Basen,rot: P).(Quelle:Kni95)
1.2 Gene
Gene sind Abschnitte auf den DNA-Strangen. Die biologische Funktion des Gens
ist dab ei inder Sequenzseiner Basenpaare ko diert. Diese Informationumfasst eine
ReihevonAnweisungenzurSyntheseeinesRNA-Molekuls,dasanschliessendentwe-
der dieSynthesevon Proteinmolekulen (z.B. Enzyme)veranlasst o der selbst in der
Zelle eineFunktion
ub ernimmt. Dieser Vorgang wird alsGenexpression b ezeichnet
(s. Abs. 1.3) [Bro93 ]. Die Entschlusselung des menschlichenGenoms brachte
ub er-
raschend zuTage, dass der Mensch
"
nur\ zwischen 25000 und 40000 Gene b esitzt.
Man war bisher von einer weit hoheren Zahl ausgegangen. Der Vergleich mit der
Zahlvonbiszu130000Proteinen,lasstdenSchlusszu,dasseinGenfurdieSynthese
mehrerer Proteinezustandig seinkann,entgegender bisherigen Vorstellung.
Die Verteilung der Gene innerhalb der DNA ist dab ei alles andere als homogen.
Da gibt es ausgedehnte Wusten, in denen sich gar keine Gene b enden (intergene
DNA), wahrend sie in anderen sogenannten Hotsp ots dicht an dicht liegen (siehe
Abb. 1.3). In hoheren Organismenkonnendiese intergenenRegionenso gross sein,
dass der Anteil der Gene an der gesamten zellularen DNA nur einige Prozent b e-
tragt.
Abb. 1.3c)veranschaulicht,dassdieentsprechendeNukleotidsequenzinlediglichei-
nemderb eidenStrangederDopp elhelixenthaltenist. EntsprechendseinerFunktion
b ei Replikationsprozessen nennt man diesen Matrizenstrang. Im Durchschnitt b e-
Abschnitte der DNA, in
c) geb en die Pfeile die
Richtungen an, in der
die biologische Informa-
tion wahrend der Gen-
expression gelesen wird.
(Quelle:Bro93)
stehteinGenauseinigenhundert(n)Basenpaaren,wasinsgesamtca.4 n
Moglichkei-
ten ergibt,von denender grosste TeiltatsachlichbiologischeInformationenenthalt.
Die DNA imZellkerndes Menschen etwaist circa3000 Mbp lang [Kni95].
2
Die funktionellenEinheitender DNAsind Basentripletts. Dieseko dierendie20 un-
terschiedlichenAminosauren, welche die Bausteine der Proteine darstellen[Lin89].
Genb ereicheaufder DNAsind durchwohldenierteStart-und Stopsequenzenmar-
kiert. Das Start-Codon b esteht fur alleGene aus den dreiBasen (A T G). Ein der
Startsequenz vorgelagerter Promotor b esteht aus einer b estimmten Basensequenz,
an die Ableseenzyme in stabiler Weise binden. Zwischen Start- und Stop-Co don
liegen die dieAminosaurenko dierendenTripletts. Interessant ist, das mehrereTri-
pletts in ein und dieselb e Aminosaure ko dieren, da es 4 3
= 64 mogliche Dreier-
Kombinationender 4 unterschiedlichenBasengibt. Dreider 64 mglichenKombina-
tionen sorgen fur das Ende der Protein-Transkription. Sie lauten UAA, UAG und
UGA.
1.3 Genexpression
Die Genexpression ist der Vorgang, der die in den Genen enthaltene Information
der Zelle zuganglich macht. Dab ei wird die DNA des Gens zuerst in RNA und
anschliessend in ein Protein
ub ersetzt. Dies wird auch als das zentrale Dogma der
Molekulargenetikb ezeichnet[Cri70].
Die erste Stufe der Genexpression, die Transkription, wird von allen Genen durch-
laufen. Wahrend dieses Prozesseswird ein RNA-Molekulsynthetisiert 3
, wob ei eine
2
Megabasenpaare
3
Ineiner ZellesindimmerBausteine b eidergenetischerSaurenvorhanden.
Stufen der Genexpression
(Quelle:Bro93)
komplementare Abschrift des Matrizenstrangs des jeweiligen Gens erfolgt.
Die zweite Stufe ist die Translation. Diese wird allerdings nur von solchen Ge-
nen durchlaufen, fur diedas Endpro dukt der Expression nicht das RNA-Transkript
selbst darstellt. Das RNA-Molekul, nun als mRNA b ezeichnet, veranlasst b ei der
Translation die Synthese eines Makromolekuls, dessen Aminosauresequenz durch
die Nukleotidsequenzder mRNAb estimmtist. Die Endpro dukte der Genexpressi-
on sind dieProteine,man sprichtvon Bioproteinsynthese.
Die Aktivitat der Gene, die durch komplexe Prozesse reguliert wird, ist demnach
auf die Expression zuruckzufuhren. Quantitative Veranderungen der Genexpressi-
on wirken sich auf das Mengenverhaltnis der resultierenden Proteine aus, was die
Entstehungeines Tumorszur Folge hab en kann.
1.4 Replikation von DNA-Molekulen
Der grundlegendeProzess b ei der Entwicklung von Organismen ist dieZellteilung;
b ei jederTeilungmusseinevollstandigeKopiealler Genehergestelltwerden. Dab ei
ist ein hohes Mass an Genauigkeit notwendig, da schon sehr geringe Fehlerquoten
(z.B. 1 Fehler pro 10000 Nukleotiden) eine b etrachtliche Anhaufung an Verande-
rungen innerhalb der Gene verursachen konnen. Dadurch b estehtdie Gefahr, dass
leb enswichtigeInformationenverlorengehen.
Aufgrund der sp eziellen Basenpaarungen dient ein DNA-Strang dem anderen als
Matrize, die Dopp elhelix kann sich folglich aus demin der Zelle vorhandenen bio-
chemischenMaterial selbst replizieren.
1.5 Der Zellzyklus
Der Zellzyklus lasst sich in vier Phasen G1, S, G2 und M unterteilen. Die ersten
drei werden zur Interphase zusammengefasst. Die M-Phase heisst auch Mitose,
die Phase der Zellteilung. Sie ist b esteht aus Prophase, Metaphase, Anaphase und
Telophase. Die Gesamtheit dieser Phase b eschreibt den zyklischen Vorgang der
Replikation der DNA und Vererbung der genetischen Information auf die To chter-
zellen.
In der Interphase liegen die Chromosomen im Zellkern in der weniger stark kon-
densierten Form vor. Die eigentliche DNA-Synthese ndet in der S-Phase statt.
G1 und G2 sind
Ub ergangsphasen zwischen der Synthesephase und der Mitose. In
der G1-Phase steigtdie biologischeAktivitatder ZellemiteinemAnstieg der RNA
Pro duktion. Nachder DNA-SyntheseinderS-Phase wird dieZelleinder G2 Phase
durchBildung vonTeilungsproteinenaufdieMitose vorb ereitet. Inder mitotischen
Prophase werden Kernmembran und Nucleolen aufgelost und der Spindelapparat
um die Zellp ole ausgebildet. Anschliessend bilden sich zwischen den Polen Spin-
delfasern, die mit einer Anordnung der Chromosomen durch den Spindelapparat
einhergeht. Die Metaphaseplatte entsteht (Metaphase). Als Anaphase b ezeichnet
man den folgenden Vorgang der Trennung der Schwesterchromosomen hin zu den
Zellp olen. In der Telophase ndet der eigentlicheTeilungsprozess statt: Kernmem-
bran und Nucleolen werden neu gebildet, die Chromosomen dekondensieren, die
Zellplatte wird gebildet, dieZelle teiltsich. Der Zellzyklusist durchlaufen und die
DNA-Synteseinder Interphase kann neu b eginnen.
DurchAufschraubungs- und Faltungsmechanismenkondensieren dieChromosomen
in der Mitoseund erreicheneinemittlereLange von 5-10 m[Czi81]. Siesindnach
Farbung im Mikroskop alsChromosomenin ihrer b ekannten Form sichtbar. Durch
den Ansatzpunktder Spindelfasern, demCentromer wird das Chromosom ineinen
kurzen p- und eine langenq-Armunterteilt.
1.6 Genetische Ver
anderungen
Der Entstehung karzinogener Zellen gehen verschiedene genetische Veranderungen
der DNA voraus, die man in zwei Klassen unterscheidet. Mutationen sind Punkt-
veranderungen, d.h.AustauscheinereinzigenBaseo derVeranderungen,diesichauf
einen sehr b egrenztenBereichder DNAb eschranken. Sie entstehenhaug wahrend
des Replikationsprozesses o der durch chemische und physikalische Mutagene. Um
die Zahl dieser oft auf naturliche Weise entstehenden Mutationen gering zu hal-
ten, existieren Reparatur-Mechanismen. Darunter sind Enzyme zu verstehen, die
b eim
"
Abfahren\derEinzelstrangedieDNAerneuern,ab er auchgegeb enenfallsden
"
programmierten Zellto d\ (Apoptose) einleitenkonnen. ImGegensatz zuMutatio-
nen b ezeichnetmanmitRekombinationen dieUmordnungenvonDNA-Abschnitten.
Diese sind zumTeil erwunscht,da auf diese Weise genetische Vielfaltentsteht und
erhalten wird.
SolchegenetischeVeranderungenkonnendieRegulationsmechanismenderartin ih-
rer Funktion storen, dass sichein ungebremstes Wachstum entwickeltund letztlich
Deletion (Verluste) und Amplikation (Vervielfaltigungen) gewisser Gensequenzen
typischeMerkmale.
1.7 In Situ Hybridisierung
Ende dersechzigerJahrewurdevonGallund PardueeineTechnikentwickelt,diees
ermoglicht,einzelneRegionenderDNAzumarkieren. DazuwirddieDNAzunachst
denaturiert, d.h. die Wasserstobruckenbindungen der Basenpaare werden aufge-
bro chenunddieDNAausihrerdopp elhelikalenStrukturindieb eidenEinzelstrange
getrennt. AuftrennenderDNAinihreEinzelstrange(Denaturierung) geschiehtz.B.
durchWarme[Str96]. Danachwird dieDNAkomplementarenDNA-Prob enwieder
renaturiert. DenVorgang der BindungeinermarkiertenDNA-Einzelstrangprob ean
denaturiertechromosomaleDNAb ezeichnetmanalsHybridisierung. EineBindung
ndet
ub erall dort statt, wo sich komplementareBasensequenzenb enden.
Im Falle der chromosomalen in-situ-Hybridisierung b endet sich die denaturierte
DNAi.A.aufeinemObjekttrager,wahrenddermarkierteStrangvoneinerzugesetz-
ten Sonde stammt. Zunachst wurden zur Markierung radioaktive Stoe verwendet,
bspw.
3
H und der Nachweis der hybridisiertenund markierten DNA erfolgte ub er
Autoradiographie. Allerdings ist dieses Signal nicht immer deutlich sichtbar und
ub erdeckt zudem einen relativgrossen Teil des Chromosoms. Bei der Fluoreszenz-
in-situ-HybridisierungsindhingegendieNukleotidemituoreszierendenSeitengrup-
p enmarkiert. DieFluoreszenzsignale,diemittelseinemFluoreszensmikroskopnach-
gewiesen werden, sind aufgrund ihrer Strahlungseigenschaften b esser geeignet; die
dadurcherreichtehohereAuosungistgegenub erdemerstgenanntenVerfahrenvon
grossem Vorteil.
Ub erdieslassen sich mit unterschiedlichmarkierten DNA-Sonden
(klonierte DNA-Fragmente)verschiedeneGenortein einemExp erimentanfarb en.
Diese Metho de machteineortsspezische Bindung einer DNA-Prob e allerdings nur
dann moglich, wenn die zum markierten DNA-Stuck komplementare Nukleotidse-
quenz nur an einem Ort eines individuellen Chromosoms vorkommt. Es handelt
sichin dieseFall umeineEinzelkopie-Seque nz. Sollte dieDNA-Prob e jedo ch neb en
ortssp ezischen Sequenzen auch ho ch- o der mittelrep etitive Sequenzen enthalten,
die auf allen Chromosomen vorkommen,hybridisiert die DNA-Prob e an alleChro-
mosomen, so dass das ortsp ezische Signal nicht langer erkennbar sein wird. Man
istaus diesemGrundindenletztenJahrenzur CISS-Hybridisierung (Chromosomal
In Situ Suppression)
ub ergegangen [Cre88]. Zu einem geeigneten Zeitpunkt wer-
den in die Hybridisierungslosung nicht markierte ho chrep etitive DNA-Sequenzen
hinzugegeb en, die an die rep etitiven Sequenzen der Chromosomen binden. Eine
unsp ezische Bindung der markierten Prob en wird damit unterdruckt. Die Tech-
nik ermoglichtnicht nur die sp ezische Markierung von Metaphase-Chromosomen,
DNA,diealleSequenzeneinesb estimmtenChromosomsenthalt(Library-DNA).Sol-
che chromosomensp ezischen DNA-Bibliothekenkonnten entwickeltwerden, nach-
dem es gelang, individuelle Chromosomen mit dem Verfahren der uoreszenzakti-
vierten Chromosomen-Sortierunganzureichern[Cre84].
1.8 Multiplex-Fluoreszenz in situ Hybridisierung
DadieWahrscheinlichkeitzurDetektiongenomischerAnomalienmitderZahlgleich-
zeitig verwendeter DNA-Prob en steigt, wurden in den letzten Jahren verschiedene
Techniken entwickelt, um moglichst viele gleichzeitig hybridisierte Prob en zu un-
terscheiden. FISH ist dab ei die ideale Basis fur diese Metho den, da eine Vielzahl
sp ektral unterschiedlicherFarbstoe fur FISHzur Verfugung stehen.
Abb.1.5: Prinzip desM-FISH Verfahrens.(Quelle:[Eil98])
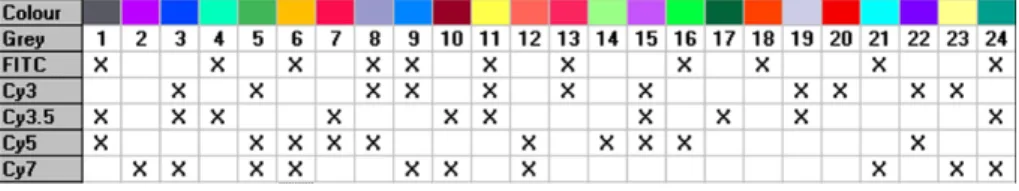
BeiM-FISHwerdenmehreresp ektralunterschiedlicheFarbstoezurMarkierung
der DNA verwendet. Dab ei wird die Zahl unterscheidbarer Prob en durch kombi-
natorische Farbung signikant erhoht. Die ersten kombinatorischen Ansatze gab
es schon Ende der achtziger, Anfang der neunziger Jahre [Ned89, Ned90, Rie92a,
Rie92b, Len93 , Pop93, Wie93]. Es wird nicht mehrnur eine einzige, sondere meh-
rere Fluoro chrome gleichzeitig an die DNA-Prob en hybridisiert, die dann an ihre
komplemetaren Sequenzen binden. Da es b ei n Farbstoen 2 n
1 moglicheKom-
bination gibt, reichen5 Farbstoe aus (2 5
1=31), umdas gesamte Genom, also
alle 24 Chromosomen unterschiedlichzu farb en und damit sp ektral unterscheidbar
furein5-Farb en Exp eriment.
Abb. 1.6: Typisches Hybridisierungsschema eines 5-Farb en m-FISH Exp eriments. Jedes
ChromosomerhalteineeindeutigeKombinationvonFarbstoenunddamiteineeindeutige
sp ektrale Signatur. Die Darstellung der Chromosomen erfolgt
ub er Falschfarb en, die in
der ob erenLeistezu sehen sind.
1.9 Comparative Genomic Hybridization (CGH)
CGH wurde zur Detektion ungewohnlicher Kopienzahlveranderungen von Sequen-
zender Nukleinsaureineinemo der mehrerenGenenentwickelt[Kal92]. Grundlage
dieser Metho de ist das FISH-Verfahren. Mit CGH kann die relative Kopienzahl-
veranderungenvon tumorsp ezischenGensequenzenalsFunktiondesOrtesder ent-
sprechendenSequenzen ineinemReferenzgenomausgedruckt werden[CGH95].
Als Prob e wird hier einerseits die gesamte DNA des Tumorgeweb es und anderer-
seits die normale Referenz-DNA verwendet, wob ei die Hybridisierung bzw. Ko-
HybridisierungaufaufgespreitetenMetaphase-Chromosomen 4
gesunderZellenstatt-
ndet. Die Tumor-DNA wird dab ei gewohnlicherweise mit grunem Fluoro chrom
gelab elt, wahrend dieReferenz-DNAmiteinemroten Fluoreszenzfarbstomarkiert
wird. Gleiche Anteile von Tumor- und Referenz-DNA
"
konkurrieren\ nun um die
gleicheZiel-DNA(imfolgendenmitTargetb ezeichnet).
AhnlichzuFISHwerdendie
FluoreszenzsignalefurjedeseinzelneTargetmiteinemFluoreszenzmikroskopgemes-
sen,imAnschlussb erechnetman dasVerhaltnisvongrunemSignal(Tumorgeweb e)
zu rotem (gesundes Geweb e). Rot-dominierte Verhaltnisse deuten auf Deletionen
und grun-dominierte auf Amplikationen in der Tumor-DNA hin [CGH95]. Abb.
1.7 zeigt eintypisches Beispiel.
DerVorteildieserMetho deb estehtdarin,dassineinemeinzigenExp erimentanhand
der Ungleichverteilungder Signale(Imbalanzen)entlangder Chromosomen-Achsen
genomweitVeranderungenin den Kopienzahleneinzelner Bereicheidentiziertwer-
den konnen. Zudem sind hier keine Vorinformationen ub er Veranderungen notig.
Durch CGH konnten insb esondere hamatologische Tumoren erfolgreich analysiert
werden(
Ub ersicht, [Lic00]).
4
Bezeichnung furdieorganisiertesteFormderChromosomenstruktur
Das Prin-
zip der
CGH.(Quelle:[Rud00])
DieseMetho dehatab er auchNachteile. SoistfureineeÆzienteAnalyseeinMinde-
stanteilvon ca. 35% an Tumorzellenin der Geweb eprob e notwendig [DuM95],was
sichzum Teiljedo ch alsproblematischerweisen kann. Auch sind diese Exp erimen-
te nur b edingt als Analysemetho den mit hohem Durchsatz geeignet, weil sowohl
die Herstellung als auch die anschliessende Auswertung manuell erfolgen mussen,
was einen hohenArb eits-und Zeitaufwand b edeutet. Desweiteren schranktdieGe-
stalt des Targets die Auosung ein. Kleinere Variationen, die durchaus fur eine
Klassizierung und Prognose von entscheidender Bedeutung sein konnen, bleib en
womoglichunentdeckt.
Physikalische Grundlagen
2.1 Mikroskopie
2.1.1 Linsen
Eine Linse ist im Prinzip eine fokussierende Blende. Licht, das durch einen Spalt
tritt, wird geb eugt. Es entsteht ein Beugungsbild mit Intensitatsmaxima und -
minima hoherer Ordnungen, deren Lage von der Wellenlange und der Geometrie
des Spalts abhangen. Durch die Form der Linse werden die optischen Weglangen
fur die Lichtstrahlen so verandert, dass dieMaxima hoherer Ordnung unterdruckt
werdenundnurfurdasHauptmaximumkonstruktiveInterferenzauftritt. DasBeu-
gungsbildeinesSpaltesistdab ei dieFouriertransformiertederSpaltfunktion,diedie
Formdes Spaltsb eschreibt. Dasleuchtetunmittelbarein,wennmansichdasInten-
sitatsprol in der Betrachtungseb ene als Summeder Schwingungszentren im Spalt
nach demHuygensschenPrinzip vorstellt. Die AmplitudenfunktionU(a;b),die das
Beugungsbild inder Bildeb eneb eschreibt,verdeutlichtdiesen Zusammenhang:
U(a;b)= Z Z
x;y
s(x;y)exp i(ax+by )
dxdy (2.1)
Die Ko ordinaten in der Blendeneb ene werden durch (x;y), die Blendenfunktion
durch s(x;y) b eschrieb en, die fur einen Spalt den Wert 1 hat; a und b sind die
raumlichen Frequenzen in der Bildeb ene. Sind (X ;Y) die Ortsko ordinaten in der
Bildeb eneind L dieBrennweitederLinse, sowiek dieWellenzahl,lassen sicha und
b schreib en als a =kX=L und b=kY=L. Die vollstandige theoretische Herleitung
dieser Formelergibt sich aus demKirchhoschen Integral Theorem.
Beim Durchgang duch das Objektiv werden aufgrund der b egrenzten raumlichen
AusdehnunghohereraumlicheFrequenzenamRandabgeschnitten. Diesfuhrtdazu,
dass das Bild eines Objekts nach Durchgang durch eine Linse einen Informations-
verlust erleidetund das Bildnichtvollstandigrepro duziert wird. Die geradefurdie
der Folge,dass das Beugungsbild an den Randernunscharfwird und verschmiert.
Die Formder b ewirkt, dass Licht,das von einemb estimmten Punktvor der Linse
ausgeht, ineinen denierten Punkt hinterder Linse geb eugt wird, und zwar genau
in den Punkt, furden die optischen Weglangen der von der Punktquelleausgehen-
den Lichtstrahlen gleich, was zu konstruktiver Interferenz fuhrt. Die normalisierte
laterale Intensitatsverteilung, die ein weit entfernter Punkt in der Fokaleb ene der
Linse erzeugtlautet:
I()=
2J
1 ()
2
(2.2)
J
1
() istdieBesselfunktionerster Ordnung, der normierteRadius mit
=kRsin; (2.3)
wob eiRderRadiusder Blende,derRaumwinkelundk =2=dieWellenzahlist.
Fur!0konvergiertderQuotient2J
1
()=gegen1/2,dergesamteTermalsogegen
1. Das Hauptmaximum heisst Airy Disk. Diese wird durch die ersten Nullstellen
der Besselfunktiondeniert,ihrRadius lasst sichmitsin=R=f schreib en als
r
Air y
=0:61
0
NA
: (2.4)
Linsen weisen Abbildungsfehler auf, die die theoretische Auosung in der Mikro-
skopie verschlechtern. Die Abbildungseigenschaften von Linsen verschlechternsich
dab ei im allgemeinen mit zunehmendem Abstand von der optischen Achse. Un-
terchromatischerAberrationverstehtmandieDisp ersion verursachteunterschiedli-
cheBrechung und damit Abbildung von LichtunterschiedlicherWellenlange. Licht
hoherer Frequenz wird starker gebro chen, damit liegt der Fokus solchen Lichts vor
langerwelligem. Der Unterschied in der Lage des Brennpunkts kann im sichtbaren
Sp ektrum bis zu einer Wellenlange b etragen. Spharische Aberrrationen sind Ab-
weichungenund Fehler in der Ob erache der Linse. Ein einfaches Beispiel ist der
Astigmatismus,derdurcheinenanisotrop enKrummungsradiusverursachtwird. Bei
mo derne Mikroskop en sind solcheAbbildungsfehlerweitgehendkorrigiert. Fureine
ausfuhrlicheBehandlung zudiesemThemamussandieser Stelleaufweiterfuhrende
Sp ezialliteratur der Optikverwiesenwerden.
2.1.2 Au
osungsverm
ogen
ZuBeginnder MikroskopiewardasAuosungsvermogenvor allemdurchtechnische
Probleme b ei der Fertigung von Linsensystemen b estimmt. Vor allem chromati-
scheundspharischeAbb erationenwarendieHauptursachen furdieBegrenzungdes
Auosungsvermogens. Heutzutage sind diese Probleme grosstenteils b ehob en, so
ner Welleneigenschaftistesnichtmoglich,dasLichtdurcheineendlichgrosseLinse
in einem b eliebig kleinen Punkt zu bundeln. Die Verteilung der Lichintensitat im
Fokushat deshalb eineendlicheGrosse.
DeniertmandieAuosungsgrenzealsdenAbstandderBilderzweierPunktquellen,
b ei dem das Maximumdes einen im ersten Minimumdes anderen Beugungsbildes
ist, so ergibt sich mit=3:832,furdieerste Nullstelleder Besselfunktion
sin= 3:832
kR
=1:22
2R
=0:61
0
nR
(2.5)
Hier ist die Wellenlange n der Brechungsindex des Mediums, in dem sich das
Objektb endetundderhalb e
OnungswinkeldesObjektivs. DasPro duktnsin
wird als die Numerische Apertur (NA) des Objektivs b ezeichnet und lasst sich
auch schreib en als NA = nR=f. R und f sind resp ektive der Radius und die
Brennweite der Linse. Fur eine hohe Auosung sind also eine kleine Wellenlange
undeinehohenumerischeAp erturerforderlich. ManverwendetdaherImmersionsole
mit einemhohen Brechungsindex (n 1:5). Bei kurzen Wellenlangen (kleiner als
450 nm)
andertsichder BrechungsindexvonGlas stark,so dass dieObjektivezwar
auf ein mono chromatisches Beleuchtungslichtkorrigiertwerden konnen,jedo ch das
breitbandige Fluoreszenzlicht nicht ho chauosend zuruck abgebildet werden kann.
DiemaximaletheoretischeAp erturb etragt4,demspharischenRaumwinkel,deren
Realisierungan praktischeGrenzenstosst. SchritteindieRichtung,dieNumerische
Ap erturoptischerSystemezuerhohen,wurdenmitderEntwicklungder4und4
ThetaMikroskopie vollzogen,dieinsb esondere Auosungsverb esserungen inaxialer
Richtung erzielen[Hel94a, Hel94b]. Die theoretischeAuosung inaxialer Richtung
konnteexp erimentellveriziertwerden. Dievorausgesagte lateraleAuosungwurde
hingegen nichterreicht.
2.1.3 Axiale Auosung
EineahnlicheDenitionfurdieaxialeAuosungergibtsichmitdemerstenMinimum
des Intensitatsprols der PSF(PointSpreadFunction) inaxialer Richtung[BW80].
z
min
= 2n
0
NA 2
(2.6)
DiequadratischeAbhangigkeitderaxialenAuosungimNennerfuhrtzudrastischer
Diskriminierung in axialer Richtung b ei Erhohung der Numerischen Ap ertur, die
exp erimentell durch die 4-Mikroskopie b estatigt werden konnte. Das Verhaltnis
von axialerzu lateralerAuosung b etragt
z
min
r
Air y
= 3:28n
NA
(2.7)
DasersteConfocal-Stage-Scanning -O ptical-S ystem furtransmittiertesundreektier-
tes inkoharentes Licht wurde b ereits 1957 von M.Minsky zum Patent angemeldet
und in den folgenden Jahren weiterentwickelt. Die biologische Anwendung konnte
durch die Entwicklung der konfokalen Laser Scanning Fluoreszenz Mikroskopie er-
heblicherweitert werden ( [Cre78],[Cox82 ],[Res85]).
Beieiner konfokalen Anordnungistder Beleuchtungs-undDetektionslichtwegsym-
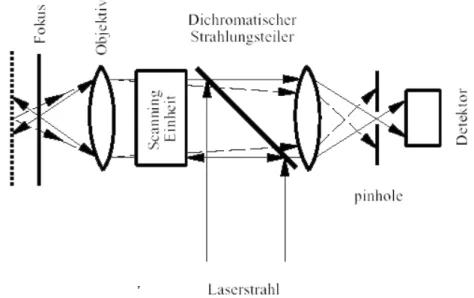
metrisch. Der prinzipielle Aufbau eines CLSM b esteht (Abb. 2.1) aus koharenter
Punktlichtquelle,Beleuchtungsobjektiv,Kollimatorlinse,Lo chblende und Detektor.
Der koharenteLichtstrahlder Laserquellewird mitHilfeeines dichroischenSpiegels
und des Mikroskop objektivs in die farbstomarkierte Prob e fokussiert; ggf. wird
hier ein erstes Pinhole zur raumlichen Filterung verwendet. Ein Teil des von der
angeregten Prob e ausgehenden Fluoreszenzlichteswird p er Objektivlinsedurch ein
Pinhole inden Detektorfokussiert.
Abb.2.1: SchematischerAufbaudes konfokalenLaser ScanningMikroskops
Als Folge der Anordnung wird eine erhohte laterale Auosung von bis zu 200 nm
und eine verb esserte Tiefenauosung von etwa 500 nm erzielt, die es ermoglicht,
dunne Schnittbildaufnahmenaus Prob en zu erhalten. Durch wiederholtes laterales
Scannen b ei verschiedenen Tiefen entlang der optischen Achse erhalt man so ein
dreidimensionalesDatenvolumen, das mit Hilfeder digitalen Bildverarb eitung aus-
gewertet werden kann.
ImVergleichzum konventionellenFluoreszenzmikroskop giltfurdie Intensitatsver-
I(z)=
2J
1 (z)
z
4
(2.8)
also im Prinzip das Quadrat von (3.4). Das liegt daran, dass sich die Punkt-
Abbildungs-Funktion von Punktlichtquelleund Punktdetektor im Fokus der Linse
ub erlagern. Das lateraleAuosungsvermogenistaufgrund der vergrossertenNume-
rischenAp erturmitderIntensitatsverteilunginobigerGleichunggegenub erkonven-
tionellen Aufnahmen um etwa 27% verb essert. Die verb esserte Auosung entlang
der optischen z-Achse ist imWesentlichen der Lo chblende zuverdanken,die direkt
vor demDetektorsystemp ositioniert wird. DurchsiewirdFluoreszenzlichtaus ob e-
ren und unteren angeregtenSchichten weitgehend diskriminiert.
DerEinsatzvonLasernalsLichtquelleinkonfokalenMikroskopieinnichtunb edingt
notwendig, hat sich ab er b ewahrt, da Laser intensives quasi-mono chromatisches
Lichtliefern,dassichgutfokussieren lasst. Damitwerden hohenPhotonenraten er-
zielt,um b eihohen raumlichenFrequenzennahe der Auosungsgrenze einenhohen
Kontrast, Signal-Rausch Verhaltnis und damit Bilder hoher Qualitat zu erhalten.
Ein Problemvon Laserlichtist seine hohe Koharenzlange,die zuInterferenzenaus-
serhalb des Fokus und in der Bildeb ene, sogenannten speckles, fuhrt, die die Bild-
qualitatb eeintrachtigen. EinPinholevordemDetektorschatAbhilfe,danurLicht
aus demFokussiervolumenin den Detektorgelangen kann.
Ein MFISH taugliches konfokales Laser Scanning Mikroskop steht no ch nicht zur
Verfugung. Mit herkommlichen CLSMs lassen sich 3-Farb en Exp erimente jedo ch
analysieren, wenn man sich auf sehr wenige Farbstoe b eschrankt. Das LEICA
TCS 4D Mikroskop b esitzt zumBeispieleinen Filterschieb erfur DAPI,FITC,Cy3
und Cy5. Diese Farbstoe lassen sichmit den vorhandenden Linien der HeNe und
Ar-Ionen Laser des Mikroskops anregen (488 und 568 nm).
Die AnalysedreidimensionalerInterphase Exp erimentemitmehrals dreiFarbstof-
fen (s. Abs. 6.6, Abb. 6.21) ist zur Zeit nur mit Epiuoreszenzmikroskopie mit
anschliessenderDekonvolutionfurjedeneinzelnenKanal moglich. Einsolches nicht
konfokales Mikroskop fur die Analyse von zweidimensionalen Aufnahmen wird in
Abschnitt2.4.1 b eschrieb en.
2.2 Laserinduzierte Fluoreszenz (LIF)
Durch Absorption eines Photons mit der Energie h kann ein Atom o der Molekul
in einen hoheren Energiezustand angeregt werden.
DieAnregungsenergie kann durch Emissioneines Photons h 0
wiederabgegeb en
werden. Als Fluoreszenz wird die spontane Emission von Strahlung b ezeichnet. Es
b esteht auch dieMoglichkeit,Molekule imoptischangeregten Niveau durch Stosse
schema fur Laser-
induzierte Fluores-
zenz.
in andere angeregte Niveaus zu bringen. In diesemFallsprichtman von Phospho-
reszenz. Bei der Fluoreszenz entsteht ein quantenmechanisch erlaubter
Ub ergang,
wahrendb eiderPhosphoreszenz einquantenmechanischverb otener
Ub ergang,mei-
stens von einemTriplett- zu einemSingulettzustand,entsteht. Dies
aussert sich in
der durchschnittlichenLeb enszeit des angeregten Zustands, dieb ei derFluoreszenz
im Bereichvon 10 8
s liegt. Typische Leb enszeiten von Phosphoreszenz liegen b ei
Millisekunden bisSekunden.
Wennjedo chdieStossaktivierungvernachlassigtwerden kann, wirdfurjedesabsor-
bierte Photon h
a
ein Fluoreszenzphoton h
Fl
(mit
Fl
a
) ausgesandt. Der
Bruchteil aller angeregten Molekule, die nicht strahlungslos deaktiviert werden,
sondern ein Fluoreszenzphoton aussenden, wird Quantenausbeute (" 1) genannt
[Dem96].
Benutzt man als Detektionsgerat einen Photomultiplier b enutzt, so erhalt man b ei
N
a
absorbiertenPhotonen innerhalb eines RaumwinkelsQ
N
e
=N
a
"(Q=4) (2.9)
Photo elektronen, diezu N
e
Signalpulsen fuhren.
e
: Sekundaremissionsko eÆzient
N
a
: Anzahl der absorbierten Elektronen
" : Quantenausb eute
ÆQ : Raumwinkel
Bei der LIF-Sp ektroskopie wird die Laserwellenlange
L
kontinuierlich durchge-
stimmt und die vom Detektor erfasste Fluoreszenzleistung P
Fl (
L
) als Funktion
von
L
gemessen. Das so erhaltene Sp ektrum nennt sich Anregungsspektrum. Die
grosste Empndlichkeitwirdfur =1, also stossfreie Bedingungen, erhalten.
Typisches Merkmal aller Farbstoe ist ein delokalisiertes -Elektronensystem, das
durch Kohlenstoringstrukturen entsteht. Die Elektronen dieses Systems konnen
leichtangeregt werden und inAbhangigkeit der Energieder emittiertenElektronen
entstehendann Farb en verschiedenerWellenlangen.
2.3 Grenzen der konfokalen Mikroskopie
In der (vor allem dreidimensionlalen) Mikroskopie gilt es, einen moglichst kleinen
Bereich eines Praparats mit einer moglichst hohen Photonendichte anzuregen, oh-
ne es dab ei zu b eschadigen, und nur aus diesem optisch angeregten Volumen eine
moglichsthohePhotonenzahl zudetektieren. DieAnzahldetektierterPhotonen pro
Pixel b etragt dab ei nur einen Bruchteil (< 1%) der erzeugten Anregungsprozesse
[Paw95]. Ein fundamentales Verstandnis dieser Prozesse ist fur einen eÆzienten
Umgang miteinemkonfokalen Mikroskop unerlasslich,um optimale Aulosung und
Bildqualitat zu erzielen. Wie sich zeigen wird, herrscht eine Art Unscharfeprinzip
b ei der Bildaufnahme,das den Anwender zwingt,einen b estmoglichenKompromiss
zwischen Auosung, Geschwindigkeit,Kontrast, Beschadigung der Prob e, Beleuch-
tungsintensitat, Quantenausb eute, sowie Sattigungs- und Bleicheekten der Farb-
stoe zunden.
2.3.1 Photonenstatistik
Um b ei einer Messung einehohe Genauigkeitzuerzielen, ist es notwendig, so viele
Photonen wie moglich zu detektieren. Photonen unterliegen der Poissonstatistik.
Die Poisson-Verteilung b eschreibt die Verteilung von unendlich vielen Messungen
seltener Ereignisse, deren Nichteintreten wesentlich wahrscheinlicher ist, als deren
Eintreten.
P(k)= n
k
k!
exp n
(2.10)
Die Wahrscheinlichkeit, dass eine gemessene Zahlrate b ei einer mittleren Zahlrate
von n Photonen zwischenn p
n und n+ p
n liegt,b etragt 63%, dieStandardab-
weichung wird allein durch den Mittelwert n b estimmt ( = n). Hat man b ei-
spielsweiseeinemittlerePhotonenzahl von100gemessen,liegen63%der Messungen
zwischen90und110. DieMessungindiesemBeispielweisteine10%ige( p
n=n)Sta-
tistik auf. Weitaus b essere Statistikenerhalt man mit hohen Photonenzahlen. Mit
n =90000 istdieStatistikauf0.3%verb essert. DieGenauigkeitderMessunghangt
von der Anzahl der gemessenenPhotonen ab. Die Photonenstatistik stellt also ei-
ne ProzessinterneRauschquelledar, diehohe Zahlraten fur zuverlassige Messungen
notwendig macht [Paw95]. Beihohen Photonenzahlen kann die Poissonverteilung
durch eineGaussverteilungangenahert werden.
2.3.2 Sattigungs- und Bleicheekte
BeinichtzustarkenBeleuchtungsintensitaten(<1mW),isteineMoglichkeit,hohere
Emissionsraten und damithohere Detektionsraten zuerhalten,dieBeleuchtungsin-
tensitat zu erhohen. Die Absorptionsrate hangt linear von der Intensitat ab, mit
der Folge, dass mehr Farbstomolekule pro Volumeneinheit angeregt werden, und
mit wachsender Besetzung angeregter Zustande steigt die Anzahl sp ontaner Pho-
tonenemissionen. DamitstimulierteEmissionsprozessedieAnregungseÆzienznicht
b eeintrachtigen,mussendieangeregten Elektronenschnelleralsdiesp ontane
Ub er-
gangwahrscheinlichkeit(10 8
s) intiefereZustande des Anregungsbandes relaxie-
ren (10 12
s), umvon dort sp ontan unter Emission eines Photons in den Grund-
zustand
ub erzugehen. Andernfalls standen moglicherweise nicht alle Atome zur
sp ontanen Emission und damit zur Detektionzur Verfugung.
BeiAnregungsintensitaten>1mW
andertsichdielineareAbhangigkeitderAbsorp-
tionsraten imFokusvolumen,da mitzunehmenderIntensitat immermehrMolekule
pro Volumeneinheitangeregt und schliesslicheinZustand erreichtwird, in dem im
Fokalvolumennichtmehrgenugend Farbstomolekulezur Anregung zur Verfugung
stehen. Da ab er die Anregungsrate von Farbstomolekulen ausserhalb des Fokus
und damit das Anregungsvolumen ansteigt, b edeutet dies eine zunehmende Ver-
schlechterung der Auosung. Wichtig ist, dass das Fluoreszenzsignal nicht mehr
nur von der Farbstokonzentration abhangt, ab er genau auf dieser Annahme baut
die Digitale Bildanalyseauf! Ein weiteres Problemergibt sich durch Bleicheekte.
Farbstomolekule verandern sich photo chemischund verlieren ihre Eigenschaft zu
uoreszieren. Wenn mit zunehmender Intensitat die Bleichrate ansteigt, wird dies
vor allem b ei Volumenaufnahmen zum Problem. Da b eim optical sectioning ein
Teil des Anregungslichtes fur jeden abgetasteten Punkt immer durch das gesamte
Praparat lauft,verschlechtertsichdiePhotonenausb euteund damitdieBildqualitat
mit zunehmenderabgetasteter Schicht.
Eineb ei Aufnahmen amMikroskopoft nichtb eachteteTatsacheist, dasses fur ein
optischesSystemmiteinerAuosung,die(unterderVoraussetzung,dassdasSystem
ab errationsfrei ist) durch die NumerischeAp ertur der Linsen, den Brechungsindex
des Mediumszwischen Objektiv und Praparat und die Anregungswellenlange de-
niert wird, nureine optimalePixel-/Voxelgrosse existiert. DiePixelgrosse istdurch
lateralenAbstand zweierb enachbarterAbtastungen gegeb en. Das Nyquist Abtast-
theoremb esagt nun,dass, umeine Struktur bestimmter Grosse auosen zukonnen,
diese Struktur 2.3 Mal abgetastet werden muss. FurdiePixelgrosse b eiAufnahmen
b edeutet das nun folgendes:
Bei gewahltem Objektiv, und damit denierter Auosung, muss das Praparat in
einem Abstand vom Auosung/2.3 abgetastet werden. Eine etwas kleinere Pixel-
grosse fuhrt in der Regel zu einer verb esserten Bildqualitat,da
Ub ergange weicher
werden, allerdings ohne Informationsgewinn. Starkes
Ub erabtasten kostet viel Zeit
und b egunstigt Sattigungs- und Bleicheekteim Praparat. Unterabtastung hinge-
gen b edeutet einenInformationsverlustund sollte vermiedenwerden. DurchUnter-
abtastung konnen Strukturenabgebildet werden, dienicht vorhanden sind. Diesen
Eekt nennt man Aliasing. Die grosste raumliche Frequenz der Aufnahme ergibt
sichaus 1/(2.3*Auosung).
2.4 Aufnahme von M-FISH-Bildern
Zwei Mikroskop-Systeme mit unterschiedlicher Detektionstechnik hab en sich am
Markt durchgesetzt. Das MFISH System von Leica Microsystems, und das SKY
System von Applied Sp ectral Imaging (ASI). Ersteres verwendet sp ezische Fil-
tersatze,umdieSp ektralinformationinjedemPixelzudetektieren. DieAnzahl der
gescannten Bildernentsprichtdab ei der Anzahl verwendeterFluoro chrome. Die im
RahmendieserArb eitanalysiertenZellenwarenausschliesslichvondiesemTyp. Das
SKYSystemb eruhtaufderFourier-Sp ektrokopie. FurjedesPixelwirddieAutokor-
relationsfunktion des Signals gemessen. Die Fouriertransformierte dieser Funktion
ergibt das gesamte Sp ektrum in jedem Pixel. Damit ist die sp ektrale Auosung
dieses Systemswesentlichhoher(bis zu5nm). BeideSystemehab en ihrevor und
Nachteile,dieimKapitel 6 (Diskussion) b espro chen werden.
2.4.1 Das MFISH System
DiesesSystem(z.B.LeicaDMRXA-RF8EpiuoreszenzMikroskop)verwendeteinen
Satzsp ezischerAnregungs-undEmissionsltersowiedichroischemStrahlteiler,die
zur automatischen Bildaufnahme auf einer motorisierten Drehscheib e aufgebracht
sind (bis zu8 Filter). Abbildung2.3 zeigtdas Prinzip der Aufnahmeschematisch.
Die Bildaufnahme einer kompletten Metaphase wird dadurch in wenigen Sekun-
den moglich. Diese Filter sind jeweils sp ezisch fur die im M-FISH Exp eriment
verwendeten Fluoro chrome, um optimale Anregungs- und Emissionsraten, b ei mi-
nimalemCrosstalk zu erzielen, da sich die Anregungs- und Emissionssp ektren der
Fluoro chromein der Regel
ub erlagern. DieseBandpasslter durfen jedo ch nichtzu
schmalbandig sein, da die Belichtungszeiten mit abnehmender Bandbreite zuneh-
men (2min)[Eil98]. FolgendeFluoro chromehab en sich zumEinsatz inM-FISH
Exp erimentenb ewahrt:
DAPI,einintensiverFarbsto, der zur Gegenfarbung der gesamten DNAver-
wendetwird, mitAbsorptions- und Emissionsmaximab ei 350 und 460nm
DEAC(426 und 480nm)
Fluorescein (FITC) (490und 520nm)
sowiedieCyanineCy3 (554 und 568nm)
Cy3.5 (581 und 588nm)
Cy5 (652 und 672nm)
Cy5.5 (682 und 703nm)
Die Bildaufnahmeerfolt
ub ereine Sensys CCD-Kamera von Photometrics (Ko-
dak KAF 1400 Chip). Standardmassig wird ein Objektiv mit 100x Vergrosserung
(Plan Ap o, NA1.4)verwendet. BeiausgedehntenMetaphasen sind 63x(Plan Ap o,
NA 1.32) Objective
ublich. Als Lichtquellen b esitzt das Mikroskop eine 75W Xe-
non Bogenlamp e und eine 100W Quecksilb erdampf-Lamp e. Tab elle (2.1) zeigt die
Sp ezikationender sp ezischenFiltersatze furausgewahlte Fluoro chrome.
Komp onente DAPI FITC Cy3 Cy3.5 Cy5 Cy5.5 Cy7
Anregungslter 36020 47515 5465.5 5805 60219 6826 74017.5
Strahlteiler 400 497 557 593 647 697 765
Emissionslter 46020 52220 5677.5 61215 66715 72020 79020
Tab.2.1: Epicub e Filterkonguration fur dasLeica DMRXA-RF8Mikroskop in nm
Die Belichtungszeiten fur die einzelnen Fluoro chrome hangen im wesentlichen von
derWahlderFiltersatzeundihrerBandbreite,derEmpndlichkeitderKamera,dem
Sp ektrum der Lichtquelle sowie dem Hybridisierungsprotokoll, also der Farb eÆzi-
enz der Prob e ab. Typische Belichtungszeiten fur die Fluoro chrome sind 0.5s fur
DAPI, 3s fur FITC, 3s fur Cy3, 0.5s fur Cy3.5, 4s fur Cy5 und 7s fur Cy7. Die
gesamteZeitfurdieAufnahmeeinerMetaphase b etragtsomit etwa20s. Mitdiesen
Belichtungszeitenwird etwadie Halfteder 12BitDynamikder CCD Kamera(4096
max. Intensitat) erreicht. Die Belichtungszeiten mit der Quecksilb erlamp esind in
der Regel kurzer als die mit der Xenon Bogenlamp e. Sie wird daher vorzugsweise
b ei Aufnahmen verwendet.
Pixelshifts konnen b ei diesemSystem durchden Wechsel der Filtersatze entstehen
und b etragen im Mittel 1-2 Pixel in x- und y-Richtung. Sie werden automatisch
durch geeigneteKorrektur-Algorithmenb eseitigt [Sp e96a ,Eil98 ].
2.4.2 Das SKY System
Das SKY System(Sp ectraCub e) b eruhtauf einemanderen Ansatz [Sch96, Gar96 ].
Statt sp ezischeFiltersatzezuverwenden,wirdmitHilfeder Fourier-Sp ektroskopie
das gesamte Sp ektrum in jedem Pixel gemessen. Dazu wird das Fluoreszenzlicht
mit Hilfeeines Kollimators ineinSagnac Interferometergefuhrt, und anschliessend
in eine CCD Camera fokussiert. Interferometer spielen eine zentrale Rolle in der
FourierSp ektroskopie. Das Prinzip wird imfolgenden erlautert.
Der einfallende Lichtstrahl wird imInterferometer in zwei koharente Strahlen auf-
geteilt, fur die eine variable optische Weglange generiert wird. Dadurch wird der
eine Strahl gegenub er der Referenzwelle in der Phase verschob en. Die koharenten
Strahlen werdenwieder vereint,dieIntensitat fur unterschiedlicheoptische Wegun-
terschiede detektiert und so das Autokorrelationssignal in jedem Pixel abgetastet.
Eine anschliessende Fouriertransformation liefert das gesuchte Sp ektrum in jedem
Pixel.
Eine(nichtmono chromatische)LichtwellemitderFeldstarkeE(t)lasstsichb eschrei-
b en durch:
E(t)= Z
k
A(k)cos(2kx !t)Æk (2.11)
Hieristk=1= dieWellenzahlund A(k)dieAmplitudederWellenkomp onentemit
der Wellenzahl k. Die mittlere Intensitat dieser Lichtwelle ist gegeb en durch das
Integral
I = Z
k A(k)
2
Æk (2.12)
DieIntensitatfurzweiinterferierendeStrahlen(derselb enLichtwelle)mitoptischem
Wegunterschied(phasenverschob ene Wellen) Lb etragt
I(L)=0:5 Z
k A(k)
2
Æk+0:5 Z
k
I(k)cos(2kL)Æk (2.13)
Der erste Summandistnichtsanderes alsdieKonstante
I. DerzweiteTermist der
Interferenzterm,derdieSp ektralinformationinFormdesoptischenWegunterschieds
L enthalt. Dieseristab er nichtsanderesalsder Realteil der Fouriertransformierten
von I(k). Die Gleichung fur diegemessene Intensitat b eim Wegunterschied L lasst
sichalso schreib enals:
I(L)=C+<(FT[I(k)]) (2.14)
Misstmandie IntensitatineinemPixelsukzessivefurunterschiedlicheL,erhalt
man ein Interferogramm(Autokorrelationsfunktion), das die Fouriertransformierte
des Sp ektrums ist. In der Praxis wird dieIntensitat fur unterschiedliche(diskrete)
Wegunterschiede gemessen, so dass das Sp ektrumin jedem Pixel dieFouriertrans-
formierteeiner diskreten FunktionI(L) ist:
I(k)= X
L
I(L)exp( i2kL)l (2.15)
Der diskreten Fouriertransformation vorgeschaltet werden eine Phasenkorrektur,
Ap o disierung und das Auullen mit Nullen. Diese Schritte sind notwendig, um
zenzu unterdrucken.
DasSagnacInterferometerbasiertaufdemPrinziprotierenderSpiegel,umeinenop-
tischen Wegunterschiedzu erzeugen. Das Prinzip rotierender (paralleler)Spiegel in
Interferometernwurde1981von[Yas81]eingefuhrt. FurkleineWinkel( <<1r ad)
ist der optischeWegunterschied demWinkelprop ortional:
L
=
c fu r <<1 r ad (2.16)
Diesp ektrale Auosungb etragt furdasSagnacInterferometeretwa0:01, also4nm
b ei 400nm. In der Praxis wird ab er mit einer wesentlich geringeren sp ektralen
Auosung gearb eitet, d.h die Intensitat I(L) wird seltener (b ei weniger optischen
Weglangen) gemessen,umdie Zeitfur dieBildaufnahmezu verkurzen.
Ublichsind
10-15nm b ei 400nm, man erhalt damit 5-10 mal soviel Farbinformation pro Pixel
wie b eim FilterbasiertenMFISH System. Die Aufnahmezeit fur ein Bild b ei einer
Auosung von 10nm b etragt ca. 60s und steigt linear mit der Auosung auf 120s
b ei 5nm an [Gar96 ].
Grundlagen der Bildverarbeitung
3.1 Klassizierung
Als Klassizierung b ezeichnet man die Zuweisung von Objekten anhand b estimm-
terMerkmalezub ekannten Klassen. Sieistnichtsp ezischfurdieBildanalyseund
ndet eine breite Anwendung auf verschiedenenGebieten. In der Bildanalyse las-
sen sich zwei Typ en von Klassizierungen unterscheiden: die Pixelorientierte und
Objektorientierte. In komplexen Fallen gelingt die Objektsegmentierungnicht mit
einem einzigen Merkmal. Dann mussen mehrere Eigenschaften verwendet werden,
sowieeinKlassizierungsprozess,der die Bildpunkteden Objektenzuweist[Jah97 ].
Falls sich die verschiedenenObjekte gut vom Hintergrund unterscheiden und sich
nicht b eruhren und ub erlapp en, ist die sehr viel einfachere objektbasierte Klassi-
kation die b essere Wahl. Der Daten- und Rechenaufwand ist sehr viel geringer,
da die auf Pixeln b eruhenden Merkmaleder Objekte
ub er das gesamte Objekt ge-
mitteltund alsObjekteigenschaftverwendetwerdenkonnen. ParameterwieGrosse
und Form,sowiedieOrientierungsind weitereEigenschaften, diealsMerkmaledie-
nen konnen. Manchmal ist auch die Kombination b eider Verfahren sinnvoll: Die
pixelbasierte zur Trennung der Objekte und anschliessend die objektbasierte zur
Klassizierung. Die Analysevon M-FISH Bildernlasst sichals Klassizierungspro-
blemverstehen. Interpretiertmandas Lab eling-Schmema(s. Abb.1.6)alsKlassen,
die die Chromosomen reprasentieren, b esteht die Hauptaufgab e darin, jedes Pixel
anhand seiner Farbinformation(Merkmale)einer dieserFarbklassen zuzuordnen.
3.2 Clusteranalyse
Dien unterschiedlichenMerkmaleeinesDatensatzes spanneneinenn-dimensionalen
Raum,denMerkmalsraumauf. JedesPixelo der ObjektwirdindiesemRaumdurch
einen Merkmalsvektor (o der auch Datum)reprasentiert. Wirdein Objekto der eine
dieser Klasse im Merkmalsraum eng b eeinander liegen. Besitzt eine Objektklasse
eine enge Verteilung im Merkmalsraum, spricht man von einem Cluster. Sind die
Cluster gut getrennt, lassen sich die Objekte gut in b estimmteKlassen separieren
(Abb.4.6). SinddieMerkmalehingegenschlecht,kann dieszufehlendeno der ub er-
lapp enden Clusternfuhren, was einefehlerfreieKlassizierungerschwert.
Verfahren zur Clusteranalyse teilen einen nicht klassizierten Datensatz in homo-
gene Cluster ein.
Ahnliche Daten werden also demselb en Cluster zugeordnet. Die
Daten werden dab ei den Clustern aufgrund von Zugehorigkeitsgraden zugeordnet.
Wahrend diese b ei deterministischenVerfahren binar sind, b eschreib ensie b ei pro-
balistischenund fuzzybasiertenMetho den dieWahrscheinlichkeitder Zugehorigkeit
eines Datums (o der Merkmalsvektors)zueinemCluster.
ZweiArtenvonClusterverfahrengiltesdab ei zuunterscheiden: die
uberwachteund
dieunuberwachteClusteranalyse. BeiersterenwirdaprioriWissenindasSystemein-
gearb eitet. Unub erwachte Verfahren hingegen clustern den Datenraum blind, und
kommenimmerdann zumEinsatz,wennkeinVorwissen
ub erdieDaten und das zu
erwartendeErgebnisvorhandenist. Isthingegen solches Wissenvorwegb ekannt, so
sollte dieses unb edingt b ei einer Clusteranalyse verwendet werden. Unub erwachte
Verfahrensindinder RegelsehrvielrechenaufwendigerundlieferninsolchenFallen
selten gleichwertigeErgebnisse.
Die Analyse der Daten mit Clusterverfahren basiert auf den Abstanden der Daten
zu den Clusterzentren, den sogenannten Centroiden. Die Cluster werden dab ei so
b estimmt,dassfuralleClusterdieSummederAbstandederPunktezumCentroiden
innerhalb eines Clusters minimal, und gleichzeitig der Abstand der Cluster unter-
einander maximal wird. Dab ei liegt die Annahme zugrunde, dass alle Daten das
gleicheGewichthab en und einemClusterzugeordnetwerden, und dass alleCluster
Daten enthalten.
Die Clusteranalyse eines Datensatzes X =fx
1
;:::;x
n
g ink Cluster lost also folgen-
des Optimierungsproblem:
C
=ar gmin
ci k
X
i=1 n
X
j=1 d
2
(c
i
;x
j
) (3.1)
Dab ei istC =fc
1
;:::;c
k
gdieMenge derCluster bzw.ihrer Centroide,und d(c
i
;x
j )
der Abstandzwischen Cluster c
i
und Datumx
j .
Dazu muss ein geeignetes Distanzmass eingefuhrt werden, das das Clusterergebnis
je nach Datenstruktur erheblich b eeinusst. Sehr haug wird die Euklidische Me-
trikverwendet,o der b eiobjektbasierten Klassizierungsproblemenaucheinbinares
Abstandsmass. Die Minimierung des Euklidischen Abstands b edeutet im
ubrigen
nichtsanderes alsdieBerechnung des Mittelwertes(Schwerpunktes).
Das b ekannteste Clusterverfahren ist der k-means Algorithmus. Er unterteilt den
Datensatz in Cluster gleicher Form, die durch das Abstandsmass b estimmt wird.
Haug wird hier der Euklidische Abstand verwendet. Das fuhrt dazu, dass der
Datenraum in hyp erkugelformige Cluster unterteilt wird. Die Cluster werden nur
durch ihreCentroidec
i
b eschrieb en. Sieb erechnen sichaus:
c
i
= P
n
j=1 x
j
n
(3.2)
Der k-meansAlgorithmusweistgute Klassikationsresultate aus, wenn dieClu-
stereine
ahnlicheFormundGrosseb esitzen,undihreFormvorderAnalyseb ekannt
ist.
3.2.2 Fuzzy-Clusteranalyse
Fuzzy-Cluster-Algorithmenzeichnensichdadurchaus, dass dieDaten den Clustern
aufgrund von Zugehorigkeitsgraden zugeordnet werden, die nicht binar sind, son-
dern Werte von 0...1 annehmen konnen. Handelt es sich um probabilistische Clu-
sterverfahren, konnen sie als Wahrscheinlichkeiten der Zugehorigkeiteines Datums
zu einemCluster interpretiertwerden. Das Optimierungsproblemin Gl.(3.1) wird
zu:
C
=ar gmin
c
i k
X
i=1 n
X
j=1 w
m
ij d
2
(c
i
;x
j
) (3.3)
Dab ei mussen diefw
ij
gfolgende Neb enb edingungenerfullen:
n
X
j=1 w
ij
> 0 8 i; i=1:::k (3.4)
k
X
i=1 w
ij
=1 8 j; j =1:::n (3.5)
Die fw
ij
g sind die Menge der Zugehorigkeitsgrade der Daten zu den Clustern,
C =fc
1
;:::;c
k
g dieMenge der Clusterbzw. ihrer Centroide, und d(c
i
;x
j
) der Ab-
stand zwischenClusterc
i
undDatumx
j
. DerExp onentm isteinFuzzier,der den
Einuss von Daten auf ClustermitgeringemZugehorigkeitsgrad b estimmt.
EineumfassendeBehandlungzumThemaFuzzy-Clusteranalysendetsichin[Hpp99].
Einige b ekannte Vertreterdieser Clusterverfahrenstelleichimfolgenden vor.
![Abb. 1.5: Prinzip des M-FISH Verfahrens.(Quelle: [Eil98])](https://thumb-eu.123doks.com/thumbv2/1library_info/5520103.1686912/21.892.368.575.506.823/abb-prinzip-des-m-fish-verfahrens-quelle-eil.webp)