AUS DEM LEHRSTUHL
Volltext
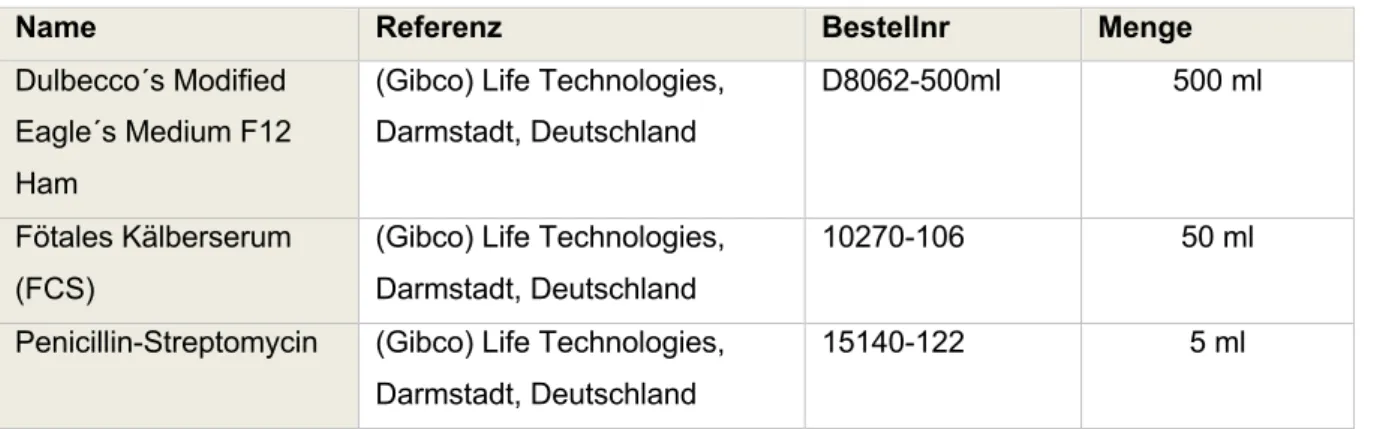
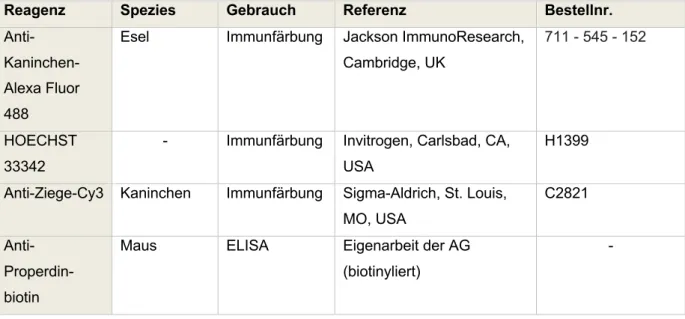
Abbildung
ÄHNLICHE DOKUMENTE
[r]
Die Dichtigkeit retrograder Wurzelfüllungen wurde in der Ver- gangenheit häufig untersucht; in der zur Verfügung stehenden Literatur konnten jedoch keine Angaben zur apikalen
[r]
87,5% der Spenderzähne wiesen zum Zeitpunkt der Operation eine noch nicht abgeschlosene und 12,5% eine abgeschlossene Wurzelformation auf.... Die besten Erfolgschancen ergaben sich
i) Dr¨ ucken Sie die drei bekannten Tatsachen mittels dieser Aussagen und logischer Verkn¨ upfungen aus.. ii) Entscheiden Sie mit Hilfe einer Wahrheitstafel,
Damit ist g auch nicht surjektiv.. Also ist h
Nachdem die Frage vorgelesen wurde, notieren die einzelnen Teams auf dem „RISIKO-Rechencenter“ ihren Antworttip und legen sich schriftlich auf die Höhe des Einsatzes
Endgültige Dimensionierung erfolgt gemäß konstruktiven und statischen Erfordernissen.