Hämodynamische Analyse und Klassifikation der Gefäßstrukturen bei Patienten mit zerebralen arteriovenösen Malformationen
Hemodynamic analysis and classification of vessel structures of patients with cerebral arterioveneous malformations
Abstract
Background:The cerebral arteriovenous malformation (AVM) is a con- genital disorder of blood vessels within the brain. An AVM represents
Nils Daniel Forkert
1Dennis Säring
1an abnormal direct connection between arteries and veins, without
Jens Fiehler
2normal capillaries between them. Thus, blood flow in other areas of the
Till Illies
2brain may decrease. Abnormal flow conditions in veins increases the risk of a hemorrhagic stroke and neurological deficit.
Dietmar Möller
3Objective: For therapy planning information about localization and
quantification of the AVM, detection of feeding arteries (Feeders) and
Heinz Handels
1draining veins, and the evaluation of the haemodynamics are required.
In this paper we present a method for the automatic detection of the 1 Institut für Medizinische Informatik,
nidus of arterioveneous malformations, its feeding arteries, draining
veins and “en-passage” vessels as well as parameters describing the Universitätsklinikum haemodynamics. Spatiotemporal 4D magnetic resonance angiography Hamburg-Eppendorf,
Deutschland (MRA) image datasets and 3D MRA datasets with high spatial resolution
were acquired for analyzing AVMs. 2 Klinik und Poliklinik für
Neuroradiologische Methods:Initially the vessel system of a 3D MRA dataset is segmented.
Then using a new method characterizing the haemodynamics by Diagnostik und Intervention, definition of a time point of inflow based on curve fitting the temporal Universitätsklinikum
Hamburg-Eppendorf, Deutschland intensity curves of 4D MRA image sequences are analyzed voxelwise.
Additionally the velocity of the blood flow is approximated. Based on a
non-linear registration method the haemodynamic information can be 3 Department Informatik, Arbeitsbereich Technische transferred automatically to the segmented vessel system. Different
Informatiksysteme, vessel structures can be characterised automatically by a combined
analysis of the intensity, velocity and a relative time point of blood inflow. Universität Hamburg, Deutschland Results:19 datasets of patients with a diagnosed AVM were available
for evaluation of the proposed method. Artefacts in terms of strong temporal leaps between the time points of inflow of two neighbouring voxels were significantly reduced after the new method extracting the time point of inflow has been applied. The automatic detection of the nidus was validated on the basis of manual segmentation. Experimental results showed a mean volume similarity of approx. 88%. Draining veins and feeding arteries were automatically detected with an accuracy of 95%.
Conclusion:The proposed method allows a robust and fully automatic detection of the AVM nidus as well as a characterization of vessels. A visual rating by neuroradiology experts showed that the proposed method describing a time point of inflow resulted in a better presentation of the blood flow than by the results achieved by the usage of conven- tional parameters. The detection of feeding arteries and draining veins is supporting the physicians in their spatial evaluation of arterioveneous malformations. The detection of the “en-passage” vessels is especially helpful for the planning of surgical resections.
Keywords:medical imaging, cerebral blood vessels, arteriovenous malformations, regional blood flow, hemodynamic analysis, registration, segmentation
Zusammenfassung
Hintergrund:Eine zerebrale arteriovenöse Malformation (AVM) ist eine Gefäßmissbildung im Gehirn, die sich durch das Fehlen eines kapillaren Gefäßbettes mit abnormem Kurzschluss zwischen dem arteriellen und dem folgendem venösen System auszeichnet, dem sog. Nidus. Die veränderten hämodynamischen Bedingungen resultieren in neurologi- schen Ausfällen sowie in dysplastischen Veränderungen der zu- und abführenden Gefäße und daraus folgenden erhöhten Blutungsrisiko.
Zielsetzung:Für die diagnostische Beurteilung der AVM sind Informatio- nen über die individuelle Gefäßstruktur und die Hämodynamik von be- sonderem Interesse. In diesem Beitrag wird ein Verfahren zur Extraktion von Parametern zur Beschreibung der Hämodynamik präsentiert. Auf- bauend hierauf werden Verfahren zur automatischen Detektion des Nidus der arteriovenösen Malformation sowie der zuleitenden (Feeder), ableitenden (Drainagevenen) und „en passage“-Gefäße vorgestellt. Als Eingabe hierfür dienen hochaufgelöste 3D- sowie zeitlich-räumliche 4D- MRT-Bildsequenzen.
Methoden:Bei der vorgestellten Methode wird zunächst in den 3D-MRT- Bilddaten das Gefäßsystem semi-automatisch segmentiert. Auf Basis eines neuen Verfahrens zur Charakterisierung der Hämodynamik durch Bestimmung des Einflusszeitpunktes des Kontrastmittels mittels refe- renzbasierter Kurvenanpassung wird in einem weiteren Schritt in den zeitlich-räumlichen MR-Bildfolgen für jedes Voxel der zeitliche Signalver- lauf analysiert. Zusätzlich wird die Flussgeschwindigkeit des Kontrast- mittels diskret approximiert. Anschließend werden die extrahierten Pa- rameterbilder mittels eines nicht-linearen Registrierungsverfahrens automatisch auf das segmentierte Gefäßsystem übertragen. Durch eine kombinierte Analyse der Intensität, der Geschwindigkeit und des relati- ven Einflusszeitpunktes des Blutes werden Gefäßstrukturen automatisch charakterisiert.
Ergebnisse:Zur Evaluation der vorgestellte Methode standen 19 Daten- sätze von Patienten mit diagnostizierter AVM zur Verfügung. Durch An- wendung der neuen Methode zur Beschreibung der Einströmzeitpunkte konnten Artefakte in Form von starken zeitlichen Sprüngen zwischen den Einflusszeitpunkten benachbarter Voxel deutlich verringert werden.
Die Detektion des Nidus wurde anhand von manuellen Segmentierungen validiert und ergab eine mittlere Volumenübereinstimmung von ca.
88%. Drainagevenen und Feeder konnten mit einer Genauigkeit von 95% detektiert werden.
Schlussfolgerung: Die vorgestellte Methode ermöglicht eine robuste automatische Detektion des AVM-Nidus sowie eine Klassifikation der Gefäße. Eine visuelle Begutachtung durch erfahrene Neuroradiologen ergab, dass bei Verwendung der vorgestellten Methode zur Charakteri- sierung des Blutflusses mittels referenzbasierter Kurvenanpassung dieser besser dargestellt werden kann, als bei der Verwendung konven- tioneller Parameter. Die Detektion von zuleitenden und ableitenden Gefäßen unterstützt den Mediziner bei der räumlichen Beurteilung der arteriovenösen Malformation. Die Detektion der „en passage“-Gefäße ist besonders hinsichtlich der Planung von neurochirurgischen Eingriffen von hoher Bedeutung.
Schlüsselwörter:medizinische Bildverarbeitung, zerebrale Blutgefäße, arteriovenöse Malformationen, Blutfluss, hämodynamische Analyse, Registrierung, Segmentierung
1 Einleitung
Die zerebrale arteriovenöse Malformation (AVM) ist eine Fehlbildung des Gefäßsystems im Gehirn. Eine AVM zeichnet sich durch ein fehlendes Kapillarbett aus, so dass in diesem Bereich das sauerstoffreiche Blut von den arteriellen Gefäßen direkt in die venösen Gefäße geleitet wird. Das Kapillarbett dient einerseits der Reduktion des Blutdruckes als auch dem Austausch von Stoffwechsel- produkten. Ein fehlendes Kapillarbett führt daher zu ei- nem anormal hohen Druck in den venösen Gefäßen.
Hieraus resultiert ein erhöhtes Risiko für eine intrazere- brale Blutung bedingt durch eine Ruptur der venösen Gefäße. In Deutschland wird pro Jahr bei ca. 800–1200 Patienten diese Erkrankung neu diagnostiziert. Das jähr- liche Risiko für eine Blutung liegt bei etwa 1–4% [1], hängt jedoch von einer Vielzahl verschiedener Risikofak- toren ab [2]. So verursacht nur etwa jede zweite AVM Letalität oder Morbidität mit bleibenden Defiziten. Es wird angenommen, dass die individuelle Anatomie und die jeweiligen hämodynamischen Verhältnisse der AVM einen entscheidenden Einfluss auf das Blutungsrisiko besitzen.
Eine verlässliche Beurteilung des Blutungsrisikos ist mit dem derzeitigen medizinischen Kenntnisstand i.A. schwer möglich.
Das vorrangige Ziel einer invasiven AVM-Behandlung ist die vollständige Ausschaltung der AVM aus dem Blutkreis- lauf. Dafür stehen u.a. endovaskuläre Embolisation, neurochirurgische Operation und stereotaktische Radio- chirurgie sowie deren Kombination als Verfahren zur Verfügung [3]. Zur Abschätzung des chirurgischen Behand- lungsrisikos wird häufig die so genannte Spetzler-Martin- Skala [4] verwendet. Anhand der Variablen AVM-Größe, Tiefe der venösen Drainage und Lokalisation werden den AVMs dabei bis zu 5 Punktwerte additiv zugeordnet. Mit ansteigender Punktzahl nehmen perioperative Morbidität und Mortalität zu. Für die Planung eines chirurgischen Eingriffs ist darüber hinaus auch die Detektion von zulei- tenden Feeder, ableitenden Drainagevenen und „en passage“-Gefäße sowie die patientenindividuelle hämo- dynamischen Gegebenheiten von besonderem Interesse.
Als „en passage“ gelten solche Gefäße, die in der näheren Umgebung zum AVM-Nidus verlaufen, jedoch keine direk- te Verbindung zu ihm besitzen.
Für die Diagnostik der AVM werden unterschiedliche bildgebende Verfahren eingesetzt. MRA-Daten ermögli- chen u.a. die Differenzierung von kleinen AVMs und die Erkennung von zuführenden und abführenden Gefäßen [5]. Zur Beurteilung der individuellen Hämodynamik ist die kontrastmittelbasierte digitale Subtraktionsangiogra- phie (DSA) derzeit der Goldstandard. Diese Technik er- möglicht die Aufnahme einer Folge von 2D-Projektionsbil- dern mit einer zeitlichen Auflösung im Subsekundenbe- reich. Neben der fehlenden Tiefeninformation stellen die verwendeten ionisierenden Röntgenstrahlen, sowie Komplikationen bei der invasiven Aufnahmeprozedur eine Gefahr für den Patienten dar. Neue nicht-invasive 4D- MRA-Aufnahmetechniken ermöglichen räumlich-zeitliche (4D) Bildinformationen und erlauben so eine Reduktion
des Risikos für den Patienten bei der bildgestützen Dia- gnostik.
Das Softwaresystem AnToNIa (Abk. f.: Analysis Tool for Neuro Imaging Data) wurde am Institut für medizinische Informatik des Universitätsklinikums Hamburg-Eppendorf zur kombinierten Visualisierung und Analyse von räumli- chen und räumlich-zeitlichen MRT-Datensätzen entwi- ckelt. Mit seiner Hilfe können nicht-invasive 3D- und 4D- MRT-Bildsequenzen kombiniert werden und anschließend die AVM qualitativ und quantitativ analysiert werden.
Die Anzahl der Veröffentlichungen, welche sich mit der Segmentierung, computergestützten Analyse und Visua- lisierung von Gefäßsystemen des Gehirns beschäftigen, ist hoch. Jedoch ist ein Segmentierungs- oder Analysetool speziell für die Problematik einer komplizierten arteriove- nösen Malformation den Autoren nicht bekannt. Bullitt et al. [6] stellen die AVM mit Volume-Rendering Technik in Kombination mit den Oberflächenmodellen der Gefäße dar und ermöglichen so eine Visualisierung der kompli- zierten Struktur. Eine Kombination mit hämodynamischen Informationen wird dort nicht beschrieben.
Um die Größe und Lage einer AVM zu bestimmen, ist eine genaue Detektion des AVM-Nidus von zentraler Bedeu- tung. In der Literatur findet sich hierfür lediglich die Arbeit von Nyui et al. [7], die auf Faktoranalyse beruht und ein Vorwissen über arterielle, venöse und Rauschsignale voraussetzt.
Für die Beurteilung der AVM sind Informationen über die individuelle Struktur und die Hämodynamik von besonde- rem Interesse. Hierzu wird im Rahmen dieses Beitrages eine Methode für die Definition eines Einflusszeitpunktes anhand zeitlicher Signalverläufe vorgestellt. Aufbauend hierauf werden Verfahren zur automatischen Detektion des Nidus der arteriovenösen Malformation (AVM) sowie der zuleitenden (Feeder), ableitenden (Drainagevenen) und „en passage“-Blutgefäße präsentiert.
2 Material und Methoden
2.1 MRA-Datensätze
Für die Entwicklung und Evaluation der beschriebenen Methoden standen 18 Datansätze von Patienten mit diagnostizierter AVM zur Verfügung. Die MR-Messungen wurden auf einem 3 Tesla Trio Scanner der Firma Sie- mens unter Verwendung einer 8-Kanal-Phased-Array- Spule aufgenommen.
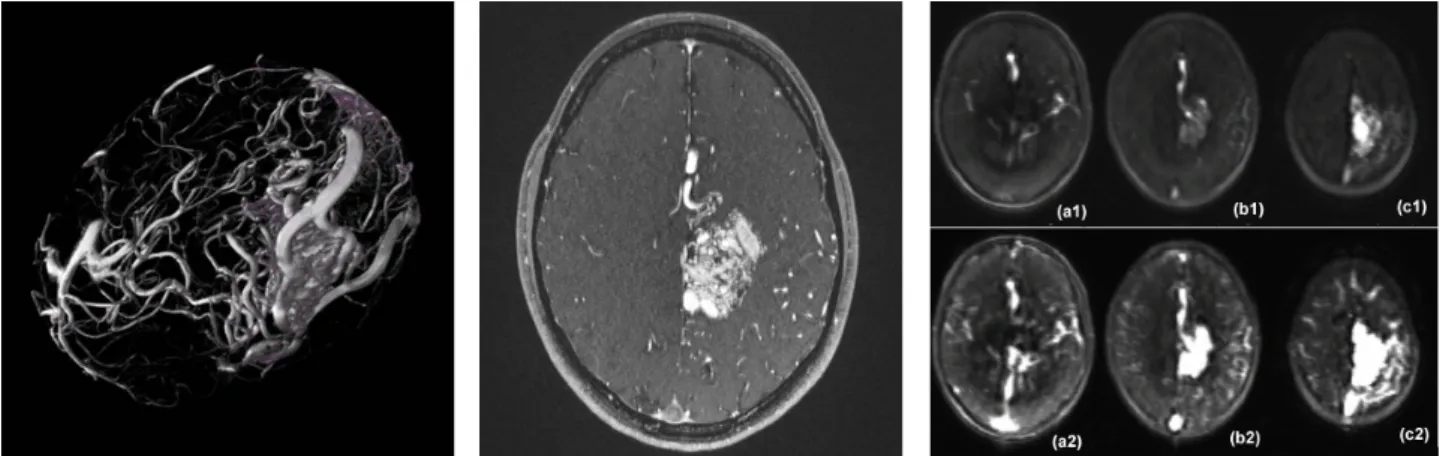
Die im Rahmen dieser Arbeit verwendeten 3D-TOF-Bildse- quenzen (Abbildung 1, Mitte) wurden mit einer Repetiti- onszeit von 23 ms, einer Echozeit von 4,09 ms, einem Flipwinkel von 18°, 5 Schichtblöcken (Slabs) mit je 40 Schichten, einer Schichtdicke von 0,5 mm und einer In- Schichtauflösung von 0,47 mm x 0,47 mm aufgenommen.
Nach einer geräteseitigen Vorverarbeitung der Slabs be- steht jeder TOF-Datensatz aus 156 Schichten. Die TOF- Bildsequenzen ermöglichen auf Grund der hohen räumli- chen Auflösung eine detaillierte Segmentierung des Ge-
Abbildung 1: 3D Gefäßsystem mit AVM (links), TOF MRT (Mitte) und 3 TREAT Schichten (a-c) mit einem zeitlichem Abstand von 6 Sekunden (rechts)
fäßsystems und eine Quantifizierung von Größe und Lage der AVM.
Im Jahr 2005 wurden die technischen Einzelheiten der
“time resolved echo shared angiographic technique”
(TREAT) publiziert [8]. Diese ermöglicht eine kontrastmit- telgestützte Datenakquisition mit einer zeitlichen Auflö- sung im Sub-Sekundenbereich. Bei dieser Technik werden parallele Bildgebungstechniken und Echo-Sharing kombi- niert verwendet, um so eine möglichst hohe zeitliche Auflösung zu gewährleisten. Die TREAT-Sequenzen bilden die Grundlage für die zeitliche Analyse der Hämodynamik.
Die im Rahmen dieser Arbeit verwendeten 4D-TREAT- Bildsequenzen (Abbildung 1, rechts) wurden mit einer Repetitionszeit von 2,54 ms, einer Echozeit von 0,69 ms, einem Flipwinkel von 20°, einer Schichtdicke von 5 mm und einer In-Schichtauflösung von 1,875 mm x 1,875 mm aufgenommen. Pro Patient standen ca. 100 3D- TREAT-Bildsequenzen mit einem zeitlichen Abstand von 0,5 Sekunden und jeweils 16 Schichten zur Verfügung.
Bei der Aufnahme der Treat-Bildsequenzen wurde das Kontrastmittel MultiHance®von Altana (20 ml) verwendet.
Die beschriebenen Methoden wurden in dem Software- prototypen AnToNIa (Analysis Tool for Neuro Imaging Data) unter Verwendung der Toolkits ITK & VTK implemen- tiert. Klassen wurden problemorientiert angepasst und eigene Klassen in C++ implementiert.
2.2 Vorverarbeitung der räumlich hochauflösenden TOF-Bildfolgen
Die Time-of-Flight Datensätze werden mit der so genann- ten Multi-Slab-Technik aufgenommen. Hieraus resultieren jedoch Schicht-zu-Schicht Amplitudenvariationen, die durch die Imperfektionen des Schichtprofils hervorgerufen werden [9]. Um diese Amplitudenvariationen auszuglei- chen, werden benachbarte Schichtblöcke mit einer Überlappung von 20 bis 30% gemessen. Im Anschluss daran werden diese Überlappungen mathematisch mittels Durchschnittsbildung oder Bestimmen des Maximums kombiniert. Trotz der gerätetechnischen Vorverarbeitung kommt es im Bereich der Überschneidungen der einzel- nen Schichtblöcke häufig zu einem Abfall der Amplitude,
was bei einer Weiterverarbeitung zu ungewünschten Er- gebnissen führen kann. Dieses Phänomen wird durch den Begriff des „Slab Boundary Artefakts“ (SBA) beschrie- ben. Zur automatischen Reduktion von Intensitätsinho- mogenitäten in MR-Bildfolgen wurde das von Kholmovski et al. [10] vorgestellte Verfahren verwendet.
Zusätzlich wurde nach Korrektur des „Slab Boundary Ar- tefakts“ die systematische Messabweichung (Bias Field) mittels der von Styner et al. [11] vorgeschlagenen Metho- de korrigiert. Abschließend wurde eine anisotrope Glät- tung [12] zur Reduktion von starkem Rauschen in den Bilddaten durchgeführt.
2.3 Kombination von TREAT- und TOF- Bildfolgen
2.3.1 Bestimmung der Einströmzeitpunkte mittels referenzbasierter Kurvenanpassung
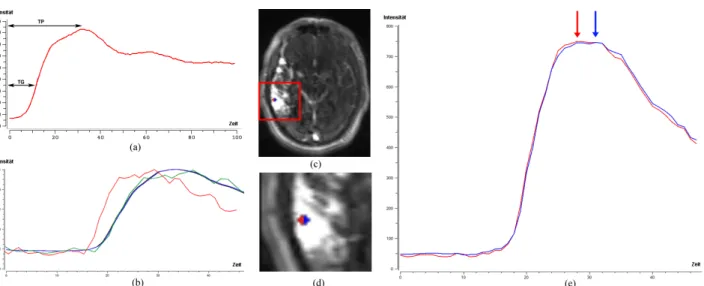
Für die Definition eines Einflusszeitpunktes für einen Kontrastmittel-Bolus anhand eines zeitlichen Signalver- laufs wurden eine Vielzahl an Methoden vorgestellt. Für einen umfassenden Überblick wird an dieser Stelle auf die Arbeit von Shpilfoygel et al. verwiesen [13]. Eine häufig verwendete Methode ist es, den Einflusszeitpunkt direkt anhand von markanten Punkten des zeitlichen Si- gnalverlaufs zu definieren. Beispiele hierfür sind “Time To Peak” (TP) und “Time to Peak Gradient” (TG) (siehe Abbildung 2a).
Diese Parameter führten bei den vorliegenden 4D-TREAT- Bildfolgen teilweise zu unrealistischen Zeitabständen zwischen den Einflusszeitpunkten zweier direkt benach- barter Voxel (siehe Abbildung 2c–e).
Daher wurde eine Methode für die Bestimmung eines Einflusszeitpunktes entwickelt, die auf der Kurvenanpas- sung eines zeitlichen Signalverlaufs und einer Referenz- kurve basiert, um so das Auftreten der beschriebenen Artefakte zu reduzieren.
Zur Analyse der Hämodynamik auf Basis der 4D-MRA- Bilddaten wird zunächst ein für Blutgefäße typischer Re- ferenzsignalverlauf patientenindividuell extrahiert. Als Referenzkurve r(t) dient eine normierte, über die Signal-
Abbildung 2: Konventionelle Parameter zur Definition eines Einflusszeitpunktes (a), zeitlicher Intensitätsverlauf (rot), Referenzkurve (blau), angepasster zeitlicher Intesitätsverlauf (grün) (b), Zwei Voxel aus einer 4D-TREAT-Schicht (c), Vergrößerung des rot umrandeten Bereichs (d), die dazugehörigen zeitlichen Intensitätsverläufe mit einem zeitlichen Abstand nach dem TP von 1,5
Sekunden (e)
verläufe aller Gefäßvoxel gemittelte Kurve. Dabei werden Signalverläufe mit atypischen Intensitätsverläufen nicht in der Berechnung mit eingeschlossen. Durch eine an- schließende B-Spline-Interpolation kann die Referenzkur- ve auch zwischen den ermittelten Signalwerten ausgewer- tet werden. Somit ist eine genauere Abschätzung des Einflusszeitpunktes möglich.
Um für einen Voxel den Einflusszeitpunkt zu bestimmen wird der dazugehörige zeitlichen Signalverlauf ѕ(t) = (ѕ1,...,ѕm) an die Referenzkurve r(t) = (r1,...,rn) linear ange- passt, so dass ||s(At + B) – r(t)||→ min gilt, wobei B die Verschiebung darstellt und A den Skalierungsfaktor. Der Faktor A dient dazu die Zeitachse zu skalieren um so die unterschiedlichen Geschwindigkeiten des Blutflusses zu berücksichtigen.
Bei der Auswahl der besten Kurvenanpassung kommt als Ähnlichkeitsmetrik die Methode der kleinsten Quadra- te zur Anwendung, so dass
gilt, wobei ƒ ein Polynom der Form ƒ(t) = At + B ist.
Durch Veränderung des Skalierungsfaktors ergibt sich ein Offset, der bei der Kurvenanpassung beachtet werden muss. Unter Verwendung des Powell-Optimierungs-Algo- rithmus [14] werden dann die Parameter A und B iterativ bestimmt, so dass E minimal wird. Der so bestimmte Parameter B wird als Einflusszeitpunkt verwendet.
2.3.2 Bestimmung der Flussgeschwindigkeit
In Anlehnung an das Maximum Slope Model [15] wird aus den zeitlichen Signalverläufen ein Parameter zur Charakterisierung der Flussgeschwindigkeit extrahiert.
Hierbei wird die Ableitung der Signalkurve unter Verwen- dung von Regressionsgeraden näherungsweise bestimmt.
Als Flussgeschwindigkeit dient das Maximum der Ablei- tung der Signalkurve.
2.3.3 Übertragung der hämodynamischen Parameter auf das Gefäßsystem
Um die zeitliche Information der Dynamik des Blutes aus den TREAT-Bilddaten und die räumliche Auflösung der TOF-Bilddaten zu kombinieren, wird zunächst für jedes Voxel der TREAT-Sequenz der Intensitätsverlauf über die Zeit analysiert. In einem weiteren Schritt werden voxelwei- se die Einströmzeitpunkte und Flussgeschwindigkeiten des Blutes nach der oben beschriebenen Methode be- rechnet, wodurch das zeitlich-räumliche Datenvolumen auf zwei 3D-Parameterdatensätze reduziert wird.
Um die TREAT- und die TOF-Bildfolgen zu kombinieren, wird in einem ersten Schritt eine 3D-Maximum Intensity Projection (MIP) über alle Zeitpunkte aus den 4D-TREAT Daten berechnet. Die 3D-MIP ermöglicht eine verbesserte Darstellung von charakteristischen Gefäßverläufen. Die daraus entstandenen zusätzlichen Bildinformationen sind hilfreich für den Registrierungsprozess. Für das hier ver- wendete nicht-lineare B-Spline 3D-3D Registrierungsver- fahren wird zunächst mittels Skalierung die Auflösung der 3D-MIP an die der 3D-TOF-MRA Bildsequenz ange- passt und anschließend die hochskalierte MIP mit dem TOF-MRA Datensatz unter Verwendung der Mutual Infor- mation als Ähnlichkeitsmaß registriert. Die aus dieser Registrierung gewonnene Transformationsmatrix kann dafür verwendet werden, die extrahierten hämodynami- schen Charakteristika auf die TOF zu übertragen.
2.4 Analyse des Gefäßsystems
2.4.1 Extraktion des AVM-Nidus
Eine Segmentierung des AVM-Nidus ist die Vorrausset- zung, um die klinisch relevanten Parameter wie Größe und Lage einer AVM sowie zuleitende und ableitende Gefäße zu bestimmen. Des Weiteren kann die Information
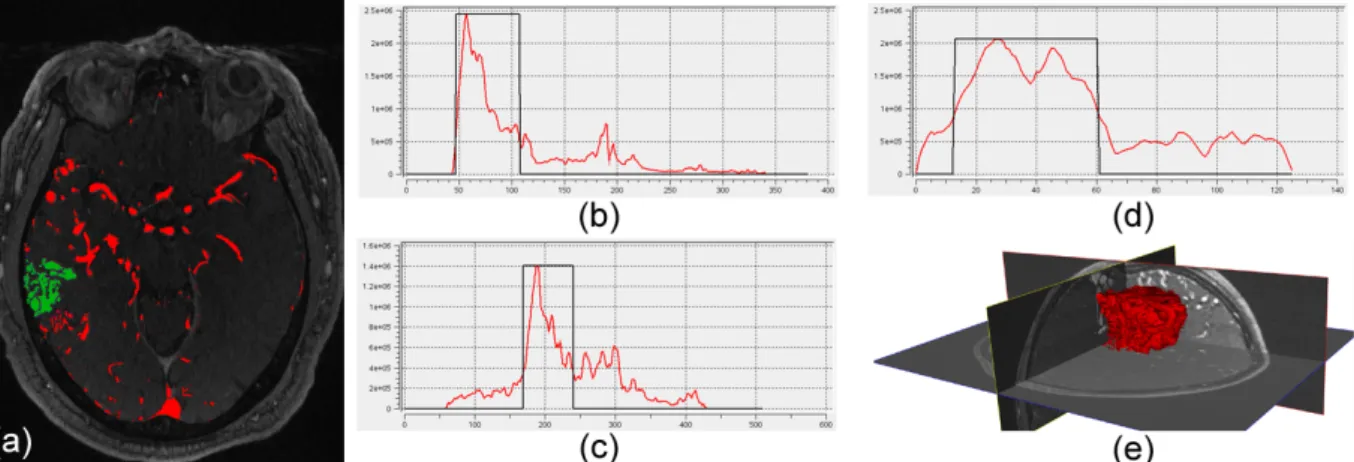
Abbildung 3: Farbcodierte Darstellung des AVM-Nidus (grün) auf TOF-MRT (a), aufsummierte P-Werte für alle Schichten in axialer, coronaler und sagittaler Ebene (b-d) und 3D-Oberächenmodell des AVM-Nidus (e)
über Lage und Größe des AVM-Nidus für eine lokale Per- fusionsanalyse verwendet werden [16].
Die hier vorgestellte Methode zur Detektion des AVM-Ni- dus basiert auf zwei medizinischen Annahmen:
1. Der AVM-Nidus stellt sich im 3D-TOF-Datensatz durch eine lokal erhöhte Dichte von hohen Intensitätswerten dar.
2. Das fehlende Kapillarbett im AVM-Nidus führt zu einer erhöhten Flussgeschwindigkeit und dadurch zu einem relativ frühen Einflusszeitpunkt des Kontrastmittels in der AVM.
Diese Eigenschaften sollen benutzt werden, um den AVM- Nidus aus den vorhandenen Bildsequenzen zu extrahie- ren.
Zur Extraktion des AVM-Nidus wird ein Parameterdaten- satz Ρ : Ω → W mit Ω Z3und W {0,1} berechnet, der unter Berücksichtigung der zuvor gemachten Annahmen die Zugehörigkeit für jedes Voxel zum AVM-Nidus charak- terisiert. Für jedes Voxel x berechnet sich P(x) durch die gewichtete Summe der Intensität I(x), der Geschwindigkeit des Blutes V(x) und dem relativen Einflusszeitpunkt T(x) (I,V,T : Ω → W):
wobei mit S(x) die Gefäßzugehörigkeit des Voxel betrach- tet wird (eine 1 stellt ein Gefäßvoxel dar).
In einem ersten Schritt wird eine Segmentierung S des individuellen Gefäßsystems aus dem 3D-TOF-Datensatz mittels Region-Growing und manueller Korrektur in ortho- gonalen Sichten generiert. Als Datensatz I dient der 3D- TOF-Datensatz, als Datensatz V der registrierte Flussge- schwindigkeitsdatensatz (siehe Abschnitt 2.2.2). Auf Basis der mittels des 4D-TREAT-Datensatzes berechneten Ein- flusszeitpunkte und der Segmentierung wird ein mittlerer Einflusszeitpunkt t’ für das Gefäßsystem bestimmt. Für jedes Voxel wird nun der Einflusszeitpunkt t von dem mittleren Einflusszeitpunkt t’ subtrahiert. So entsteht ein 3D-Datensatz T mit relativen Einflusszeitpunkten.
Die Kombination der einzelnen Parameterbilder liefert ein Datensatz, in dem sich der AVM-Nidus durch eine lo- kale Anhäufung hoher P-Werte darstellt. Um den Nidus
hieraus zu extrahieren werden die auftretenden P-Werte für jede Schicht in axialer Richtung aufsummiert. In einem weiteren Schritt werden dann Mittelwert und Maximum bestimmt. Dieses wird analog für die koronaren und sa- gittalen Ebenen durchgeführt. Ausgehend von den Mittel- werten kann eine Boundingbox berechnet werden, in der sich der AVM Nidus befindet (Abbildung 3). Neben dem AVM-Nidus können sich in der so bestimmten Bounding- box auch noch weitere Gefäße befinden, die innerhalb der Bounding Box keine Verbindung zu diesem aufweisen.
Um diese auszuschließen wird in einem abschließenden Schritt eine Zusammenhangsanalyse durchgeführt, wobei die größte Komponente den AVM-Nidus darstellt.
2.4.2 Definition von Drainagevenen, Feeder und „en-passage“-Gefäßen
Auf Basis des extrahierten Nidus kann mit Hilfe der übertragenen Einflusszeitpunkte ein mittlerer Einflusszeit- punkt des Nidus bestimmt werden. In einem weiteren Schritt werden die Gefäßvoxel in der näheren Umgebung zum AVM-Nidus analysiert. Gefäßteile, die keine Verbin- dung zum AVM-Nidus aufweisen, werden als „en passage“
eingeordnet. Für die verbliebenen Gefäßvoxel werden die dazugehörigen Einflusszeitpunkte des Kontrastmittels analysiert. Voxel, die einen früheren Einflusszeitpunkt aufweisen als der AVM-Nidus, werden als Feeder einge- ordnet, Voxel die einen späteren Einflusszeitpunkt aufwei- sen als Drainagevenen. In einem Nachverarbeitungs- schritt werden einzelne Voxel, die falsch klassifiziert wurden, durch eine Nachbarschaftsanalyse detektiert und korrigiert.
3 Ergebnisse
Bei der Entwicklung der Analyse und Visualisierungstech- niken von AnToNIa standen 18 Datensätze von Patienten mit einer diagnostizierten AVM zur Verfügung. Zur Evalua- tion erster Ergebnisse wurde in den TOF-MRT-Bilddaten aller Patienten das Gefäßsystem segmentiert und ein Oberflächenmodell erzeugt. Anschließend wurden in 4D-
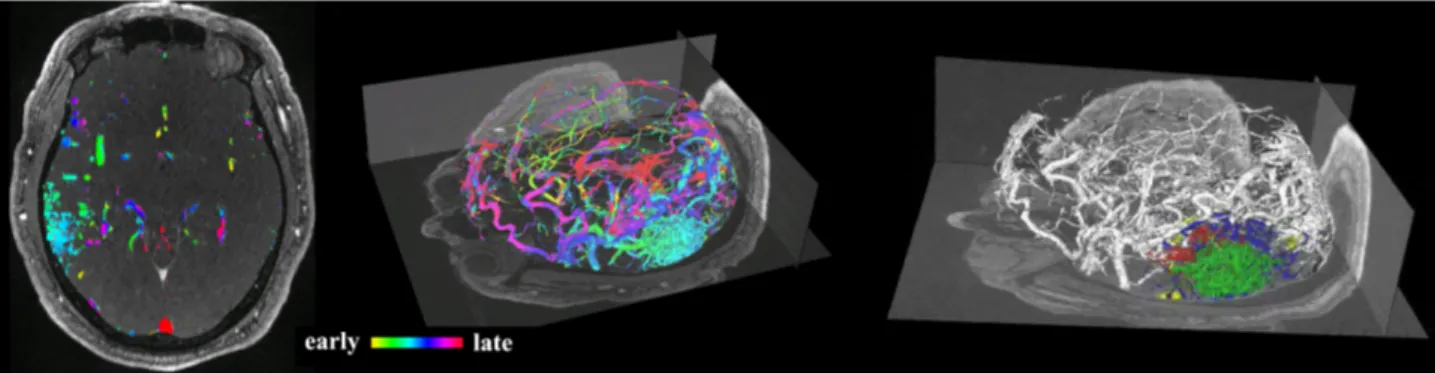
Abbildung 4: TOF-Schicht mit Einflusszeitpunkten farbüberlagerter Segmentierung (links) , Oberflächenmodell mit farbkodierten Einflusszeitpunkten (mitte), farbkodierte Darstellung der Feeder (rot), Drainagevenen (blau) des AVM Nidus (grün) sowie „en
passage“-Gefäße (gelb) auf 3D-Oberflächenmodell (rechts)
TREAT-Datensätzen Einströmzeitpunkte und Flussge- schwindigkeiten berechnet und auf Basis der vorgestell- ten Methode zur Kombination der TOF- und TREAT-Daten- sätze auf das 3D-Oberflächenmodell und auf die 2D-TOF- Schichtbildern übertragen. Dort können die extrahierten hämodynamischen Informationen farbkodiert dargestellt werden (Abbildung 4).
Durch die hier vorgestellte Methode zur Bestimmung der Einflusszeitpunkte durch Kurvenanpassung ist es im Vergleich zur Verwendung von konventionellen Parame- tern möglich, die zeitliche Auflösung zu steigern. Wesent- licher Vorteil der Methode ist, dass die Anzahl von starken zeitlichen Sprüngen zwischen den Einflusszeitpunkten zweier benachbarter Voxel deutlich verringert werden konnte. Eine visuelle Begutachtung durch erfahrene Neuroradiologen ergab, dass bei Verwendung der vorge- stellten Methode zur Charakterisierung des Blutflusses dieser qualitativ besser dargestellt werden kann als bei der Verwendung konventioneller Parameter. Die Laufzeit der hier vorgestellten Methode ist ca. 5-mal höher als die zur Extraktion der konventionellen Parameter.
Zur Evaluation der automatischen Detektion der AVM- Nidi wurden diese in den zur Verfügung stehenden Daten- sätzen von Neuroradiologen manuell segmentiert. Erste Ergebnisse der automatischen Detektion im Vergleich zu der manuellen Segmentierung ergab eine durchschnittli- che Volumenübereinstimmung von 88% und eine Sensi- tivität von 76%, bei w1, 2, 3= 1. Die Ergebnisse der automa- tischen Detektion wurden von den medizinischen Exper- ten als qualitativ wertvoll eingestuft. Die benötigte Zeit für die automatische Detektion lag bei ca. 5 Minuten, wobei bei der vorgestellten Methode keine Benutzinter- aktion von Nöten ist, im Vergleich zu durchschnittlich 30 Minuten, die für die manuelle Segmentierung benötigt wurde.
Für die Beurteilung der automatischen Detektion von Drainagevenen und Feeder wurden diese in einem ersten Schritt durch Experten, auf Basis von medizinischem Wissen bestimmt und die Ergebnisse mit der automati- schen Detektion verglichen. Dabei zeigte sich, dass durch die vorgestellte Methode eine Genauigkeit von 95% er- reicht wurde. Die Ergebnisse der automatischen Detektion von AVM-Nidus, Drainagevenen, Feeder und „en passa- ge“-Gefäßen können ebenfalls farbüberlagert in den 2D-
TOF Schichtbildern und auf einem 3D-Oberflächenmodell des Gefäßsystems dargestellt werden (Abbildung 4).
Die interaktive Navigation im 3D-Raum, Rotation und Zooming, sowie das optionale Ein- und Ausblenden der Zeitinformation von Gefäßstrukturen trägt wesentlich zur diagnostischen Unterstützung bei der Beurteilung von zerebralen Gefäßmissbildungen bei. Die kombinierte Darstellung von Anatomie und Hämodynamik erlaubt einen Informationsgewinn gegenüber der Diagnostik mittels DSA und MRA bzw. CTA.
4 Diskussion
Es wurden neue Verfahren zur Visualisierung und Analyse von AVM präsentiert. Hierbei wurden aus zeitlich-räumli- chen 4D-TREAT-Datensätzen Parameter für die Hämody- namik extrahiert und nach nicht-linearer Registrierung mit räumlich hochaufgelösten 3D-TOF-Bilddaten kombi- niert visualisiert und analysiert. Dabei können die extra- hierten Parameter optional als starre Farbüberlagerung in den TOF-Schichten und im 3D-Gefäßmodell dargestellt werden. Weiterhin wurde ein Verfahren zur Analyse der Hämodynamik präsentiert. Hierbei wurde mittels Kurven- anpassung ein gegebener zeitlicher Signalverlauf an eine Referenzkurve angepasst. Geschwindigkeitsunterschiede des Blutflusses wurden durch die Skalierung der Kurven mit berücksichtigt.
Es wurde ein neues Verfahren zur Detektion des AVM- Nidus sowie der zuleitenden, ableitenden und „en passa- ge“-Blutgefäße präsentiert. Eine erste Evaluation der automatischen Detektion des AVM-Nidus ergab eine Übereinstimmung des Zielvolumens von ca. 88% im Ver- gleich zu der manuellen Segmentierung. Um fundierter Aussagen über die Genauigkeit der vorgestellten Methode machen zu können, sind manuelle Segmentierungen von weiteren Experten im Bereich der Neuroradiologie notwen- dig, da auf Grund der hohen Komplexität der Struktur der AVM mit einer hohen Inter-Observer-Varianz zu rechnen ist. Die durchschnittliche Zeit von ca. 5 min, die für die automatische Detektion benötigt wird und währenddes- sen keine Benutzerinteraktion von Nöten ist, stellt im Vergleich zu den 30 min, die im Mittel für die manuelle Segmentierung benötigt wird, eine deutliche Zeitersparnis
dar. Die Detektion von zuleitenden und ableitenden Ge- fäßen wurden von erfahrenen Neuroradiologen als hilf- reich insbesondere für die Diagnose und Therapieplanung eingestuft.
Für die nähere Zukunft ist geplant, die Laufzeit des Algo- rithmus zur Bestimmung der Einflusszeitpunkte zu verrin- gern. Zusätzlich soll die vorgestellte Methode zur Defini- tion von Einflusszeitpunkten quantitativ anhand von Flussphantomen evaluiert werden.
Anmerkungen
Interessenkonflikte
Keine angegeben.
Danksagung
Diese Arbeit wurde von der Deutschen Forschungsgemein- schaft gefördert (Ha2355/10-1).
Literatur
1. Choi JH, Mohr JP. Brain arteriovenous malformations in adults.
Lancet Neurol. 2005;4(5):299-308. DOI: 10.1016/S1474- 4422(05)70073-9
2. Al-Shahi R, Warlow C. A systematic review of the frequency and prognosis of arteriovenous malformations of the brain in adults.
Brain. 2001;124(10):1900-26. DOI:
10.1093/brain/124.10.1900
3. Grzyska U. Hamburger Konzept bei zerebralen arteriovenösen Malformationen [Treatment of cerebral arteriovenous malformations: Hamburg Concept]. Clin Neuroradiol.
2004(1);14:41-7. DOI: 10.1007/s00062-004-5375-5 4. Spetzler RF, Martin NA. A proposed grading system for
arteriovenous malformations. J Neurosurg. 1986;65(4):476-83.
DOI: 10.3171/jns.1986.65.4.0476
5. Fasulakis S, Andronikou S. Comparison of MR angiography and conventional angiography in the investigation of intracranial arteriovenous malformations and aneurysms in children. Pediatr Radiol. 2003;33(6):378-84.
6. Bullitt E, Aylward S, Bernard EJ, Gerig G. Computer-assisted visualization of arteriovenous malformations on the home personal computer. Neurosurgery. 2001;48(3):576-82. DOI:
10.1097/00006123-200103000-00024
7. Nyui Y, Ogawa K, Kunieda E. Extraction of arteriovenous malformation with factor analysis. Image Processing, 2000 Proceedings. 2000;2:621-4. DOI: 10.1109/ICIP.2000.899514 8. Fink C. Ley S, Kroeker R, Requardt M, Kauczor MU, Bock M. Time-
resolved contrast-enhanced three-dimensional magnetic resonance angiography of the chest: combination of parallel imaging with view sharing (TREAT). Invest Radiol. 2005;40(1):40- 8.
9. Laub G, Gaa J, Drobnitzky M. Techniken Der Magnetresonanz- Angiographie. Electromedica. 1998;66(2):62-70.
10. Kholmovski E, Alexander A, Parker D. Correction of slab boundary artifact using histogramm matching. J Magnetic Resonance Imaging. 2002;15:610-7. DOI: 10.1002/jmri.10094 11. Styner M, Gerig G, Brechbuehler C, Szekely G. Parametric
estimate of intensity inhomogeneities applied to MRI. IEEE Transaction On Medical Imaging. 2000;19(3):153-65. DOI:
10.1109/42.845174
12. Perona P, Malik J. Scale-space and edge detection using anisotropic difusion. IEEE Transaction On Pattern Analysis Machine Intelligence. 1990;12(7):629-39. DOI:
10.1109/34.56205
13. Shpilfoygel SD, Close RA, Valentino DJ, Duckwiler GR. X-ray videodensitometric methods for blood flow and velocity measurement: a critical review of literature. Med Phys.
2000;27(9):2008-23. DOI: 10.1118/1.1288669
14. Press WH, Teukolsky SA, Vetterling WT. Numerical Recipes in C.
2nd ed. Cambridge: University Press; 1993.
15. König M, Klotz E, Heuser, L. Zerebrale Perfusions-CT – Theoretische Grundlagen, methodische Realisierung und praktische Erfahrungen in der Diagnostik des ischämischen Hirninfarktes. Fortschr Rontgenstr. 2000;172(3):210-8. DOI:
10.1055/s-2000-109
16. Säring D, Forkert ND, Fiehler J, Piening M, Handels H. Analyse des zerebralen Blutflusses bei arteriovenösen Malformationen unter Verwendung von 3D MRA und ASL Bilddaten. In: Kongress Medizin und Gesellschaft 2007; Augsburg, 17.-21.09.2007.
Düsseldorf: German Medical Science GMS Publishing House;
2007. Doc 07gmds674 Available from:
http://www.egms.de/en/meetings/gmds2007/07gmds674.shtml
Korrespondenzadresse:
Nils Daniel Forkert
Institut für Medizinische Informatik, Universitätsklinikum Hamburg-Eppendorf, Martinistraße 52, 20246 Hamburg, Deutschland
n.forkert@uke.uni-hamburg.de
Bitte zitieren als
Forkert ND, Säring D, Fiehler J, Illies T, Möller D, Handels H.
Hämodynamische Analyse und Klassifikation der Gefäßstrukturen bei Patienten mit zerebralen arteriovenösen Malformationen. GMS Med Inform Biom Epidemiol. 2009;5(3):Doc19.
Artikel online frei zugänglich unter
http://www.egms.de/en/journals/mibe/2009-5/mibe000098.shtml Veröffentlicht:04.08.2009
Copyright
©2009 Forkert et al. Dieser Artikel ist ein Open Access-Artikel und steht unter den Creative Commons Lizenzbedingungen
(http://creativecommons.org/licenses/by-nc-nd/3.0/deed.de). Er darf vervielfältigt, verbreitet und öffentlich zugänglich gemacht werden, vorausgesetzt dass Autor und Quelle genannt werden.