ßioinformatische Analyse und Annotation eines eukaryotischen Genoms am Beispiel der Pflanze Arabidopsis thaliana
Ch r is t in e Sc h ü l l e r, Bio m a x In f o r m a t i c s, Ma r t in s r i e d
Kl a u s Ma y e r, G S F Fo r s c h u n g s z e n t r u mf ü r Um w e l tu n d Ge s u n d h e i t, Ma r t in s r i e d Ha n s-We r n e r Mew e s, G S F Fo r s c h u n g s z e n t r u mf ü r Um w e l tu n d Ge s u n d h e i t, Ma r t i n s r i e d
Abstract
The small weed Arabidopsis thaliana is the first plant whose genome will be completely deciphered. Since 1996, an international consortium o f scientists has been working on the sequencing and the subsequent bioinformatic analysis and annotation o f the genomic data.
Genome analysis includes the identification o f all genes and other elements on the genomic DNA and the elucidation o f possible biological roles o f the encoded proteins. The development o f appropriate tools fo r the analysis and construction o f databases to store, handle and display the huge amount o f information is a necessary prerequisite fo r such publicly funded genome projects as well as other large scale sequencing efforts in academics
or the biotech industry.
1 Einführung
Arabidopsis thaliana, ein Unkraut aus der Familie der Kreuzblütler, hat eine lange Geschichte als bevorzugter Modellorganismus in der molekularen Pflanzenbiologie. Da Arabidopsis darüber hinaus ein für Pflanzen relativ kleines Genom von ca. 130 Mb besitzt, war es das ideale Objekt für die erste vollständige Entschlüsselung der Erbinformation einer Pflanze.
Ziel des Projektes ist es, alle Gene von Arabidopsis zu verstehen. Dies ist die einzige Möglichkeit herauszufinden, was eine Pflanze zu einer Pflanze macht. Ein tieferes Verständnis der Biologie höherer Pflanzen ist dringend erforderlich, um den Herausforderungen, vor die welche Landwirtschaft in den kommenden Jahren gestellt ist, wie verbesserte Pflanzenproduktivität, Anpassungsfähigkeit und Verarbeitbarkeit, gewachsen zu sein. Arabidopsis eignet einerseits als Modell für das Verständnis der Biologie von Pflanzen im allgemeinen und als Referenzorganismus für die vielfältige Gruppe der dikotylen Blütenpflanzen im besonderen, insbesondere der verwandten Brassica-Arten wie z.B. Soja, die als landwirtschaftliche Nutzpflanzen große Bedeutung haben.
2 Das Arabidopsis thaliana Genomprojekt
Früher als in anderen wirtschaftlich wichtigen Pflanzen, wie z.B. Mais, Reis, Weizen oder Soja wurden von dem vergleichsweise kleinen Arabidopsis Genom genetische und physikalische Karten erstellt und das gesamte Genom repräsentierende Klon-Bibliotheken etabliert. Dies war eine Voraussetzung für den Beginn der systematischen Sequenzierung.
1994 startete ein europäisches Pilotprojekt und 1996 wurde die weltweite „Arabidopsis Genome Initiative“ (AGI) gegründet, an der sich Institute in Japan, den USA und Europa beteiligten. Das europäische ESSA-Konsortium („European Scientists Sequencing Arabidopsis“) umfasst mehr als 20 Labors in denen die Sequenzierung durchgeführt wurde, während die bioinformatische Sammlung und Auswertung der Daten zentral bei MIPS („Munich Information Center for Protein Sequences“) am MPI für Biochemie in München erfolgte.
194
3 Spezifische Herausforderungen bei der Genomanalyse von höheren Eukaryonten Die Genomanalyse umfasst die Identifizierung und Charakterisierung aller auf der genomischen DNA codierten genetischen Elemente, wie regulatorische Sequenzen, strukturelle Elemente und, als wichtigsten Bestandteil, alle Gene, die daraus resultierenden Proteine und deren potentielle Funktion. Prinzipiell gibt es dafür zwei Ansatzpunkte, einerseits die Identifizierung über Homologien zu bereits bekannten Genen und Proteinen (extrinsische Methode) und zum anderen die Anwendung von Algorithmen, die in der Lage sind, codierende Bereiche in der DNA zu erkennen (intrinsische Methode). Dabei ist zu beachten, dass anders als bei niederen Organismen (Bakterien, Viren), die Gene höherer Organismen häufig in Einzelteile aufgespalten sind, was die korrekte Vorhersage der Gasamtstruktur erheblich erschwert.
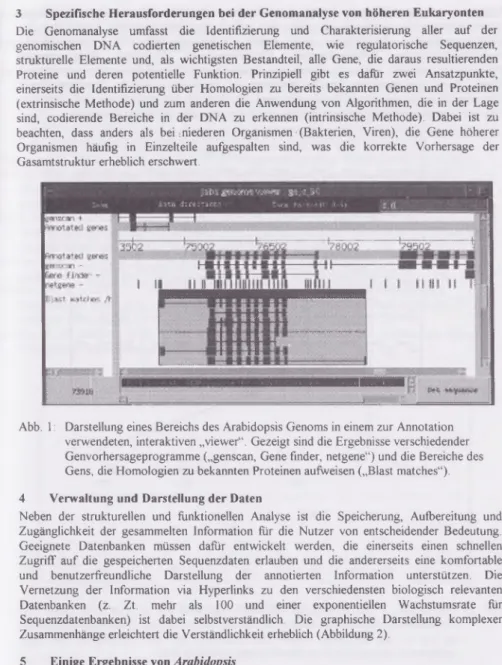
Abb. 1: Darstellung eines Bereichs des Arabidopsis Genoms in einem zur Annotation verwendeten, interaktiven „viewer“. Gezeigt sind die Ergebnisse verschiedender Genvorhersageprogramme („genscan, Gene finder, netgene“) und die Bereiche des Gens, die Homologien zu bekannten Proteinen aufweisen („Blast matches“).
4 Verwaltung und Darstellung der Daten
Neben der strukturellen und funktionellen Analyse ist die Speicherung, Aufbereitung und Zugänglichkeit der gesammelten Information für die Nutzer von entscheidender Bedeutung.
Geeignete Datenbanken müssen dafür entwickelt werden, die einerseits einen schnellen Zugriff auf die gespeicherten Sequenzdaten erlauben und die andererseits eine komfortable und benutzerfreundliche Darstellung der annotierten Information unterstützen. Die Vernetzung der Information via Hyperlinks zu den verschiedensten biologisch relevanten Datenbanken (z. Zt. mehr als 100 und einer exponentiellen Wachstumsrate für Sequenzdatenbanken) ist dabei selbstverständlich. Die graphische Darstellung komplexer Zusammenhänge erleichtert die Verständlichkeit erheblich (Abbildung 2).
5 Einige Ergebnisse von Arabidopsis
Bisher wurden zwei der fünf Chromosomen mit einer Länge von ca. 20 Mb (Chr.2) bzw. 17 Mb (Chr.4) entziffert. Dabei wurden ca. 8000 der geschätzten 22000 bis 25000 Gene von Arabidopsis identifiziert. Neben den ca. 10% bekannten Genen konnte für ca. 50% der Gene auf Grund von Homologievergleichen die vermutliche Struktur und Funktion ermittelt werden. Weitere 40% sind neue Gene, deren wahrscheinliche Struktur durch bioinformatische
195
Methoden vorhergesagt werden konnte, deren biochemische Rolle, die sie in dem Organismus spielen, aber völlig unbekannt ist. Dies sind jedoch die interessantesten Kandidaten für pflanzenspezifische Funktionen und daher Ausgangspunkte für weitergehende molekularbiologische Untersuchungen. Charakteristische Eckdaten dieser ersten Pflanzenchromosomen, wie u. a. Gendichte, Ausmass der Clusterbildung von verwandten Genen, Anteil von repetitiven Strukturelementen und die Veränderung der Verteilung dieser Elemente über das gesamte Chromosom etwa im Bereich von Centromer und Telomer, erlauben interessante Spekulationen über die Evolution von eukaryotischen Genomen.
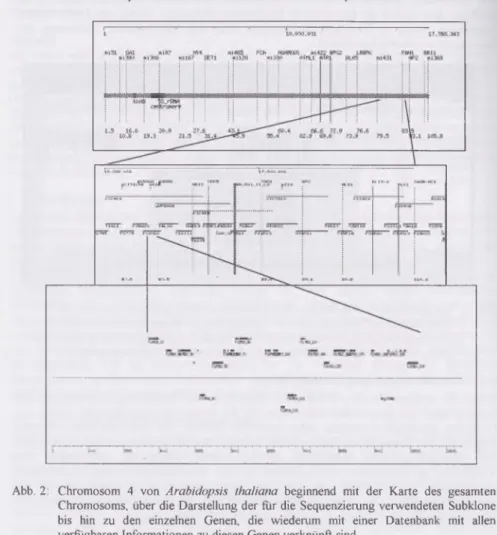
Abb. 2: Chromosom 4 von Arabidopsis thaliana beginnend mit der Karte des gesamten Chromosoms, über die Darstellung der für die Sequenzierung verwendeten Subklone bis hin zu den einzelnen Genen, die wiederum mit einer Datenbank mit allen verfügbaren Informationen zu diesen Genen verknüpft sind.
6 Literatur
B e v a n , M .; B a n c r o f t , I.; M e w e s , H.W.; M a r t i e n s s e n , R.; M c C o m b ie , R. (1999): C learing a path th ro u g h th e ju n gle: p r o g r ess in A ra b id o p sis g e n o m ic s. B io e s s a y s 21(2): 110-20.
196
Ill v a n , M.; B a n c r o f t , I.; . . . S c h u e l l e r , C.; a n d C h a l w a t z i s , N. (1998): Analysis of 1,9 Mb of contiguous sequence from chromosome 4 of Arabidopsis thaliana. Nature 391: 485-488.
lit JRSET, M.; Guigo, R. (1996): Evaluation of gene structure prediction programs. Genomics.
34(3):353-367.
Ti ih E u r o p e a n U n i o n A r a b i d o p s i s G e n o m e S e q u e n c i n g C o n s o r t i u m : K. M a y e r , C.
S c h u e l l e r , ...,C . B i e l k e , D . F r is h m a n , D . H a a s e , K. L e m c k e , H .W . M e w e s , S. S t o c k e r ,
P. Z a c c a r i a , a n d M . B e v a n a n d T h e C o l d S p r in g H a r b o r , W a s h i n g t o n U n i v e r s i t y in S t L o u i s a n d PE B i o s y s t e m s A r a b i d o p s i s S e q u e n c i n g C o n s o r t i u m : R.K. W i l s o n , ..., AND W.R M c C o m b ie (1999) S e q u e n c e an alysis o f c h r o m o s o m e 4 o f th e plant A ra b id o p sis thaliana. N a tu r e , v o l. 402, 769-777.
I r is h m a n , D.; M e w e s , H.W. (1997): PEDANTic genome analysis. Trends Genet. 13:415-6.
M e w e s , H .W .; F r is h m a n , D.; G r u b e r , C.; G e i e r , B.; H a a s e , D.; K a p s , A.; L e m c k e , K.;
M a n n h a u p t , G; P f e i f f e r , F.; S c h u e l l e r , C.; S t o c k e r , S.; W e i l , B. (2000): M IP S: a database for genomes and protein sequences. Nucleic Acids Research. 28(l):37-40.
P a v y , N.; R o m b a u t s , S.; D e h a i s , P.; M a t h e , C.; R a m a n a , DVV.; L e r o y , P.; R o u z e , P. (1999):
Evaluation of gene prediction software using a genomic data set: application to Arabidopsis thaliana sequences. [Article] Bioinformatics. 15(11) 887-899.
W a m b u t t , R ;... M a y e r , K.; S c h u e l l e r , C.; B e v a n , M . (2000): Progress in Arabidopsis genome sequencing and functional genomics. Journal of Biotechnology 78: 281 - 292.
T k r r y n , N.; H e ij n e n , L;. . . S c h u e l l e r , C ;... Vos, P. (1999): Evidence for an ancient chromosomal duplication in Arabidopsis thaliana by sequencing and analyzing a 400-kb contig at the APETALA2 locus on chromosome 4. FEBS Letters 445: 237-245.
ZACCHARIA, P.; M e w e s , H.W. (1999): Homology based gene prediction in Arabidopsis thaliana Proceedings of the German Conference on Bioinformatics.
197