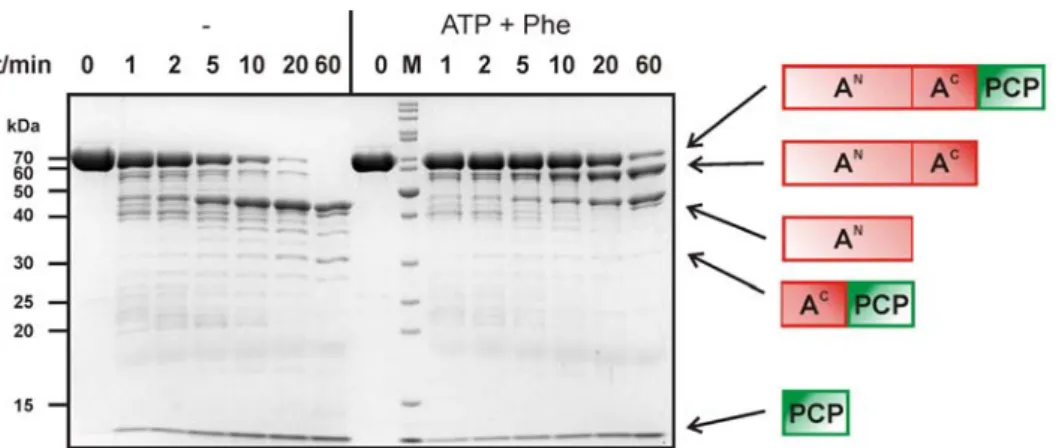
Wechselwirkungen zwischen Adenylierungs- und Peptidyl Carrier Protein-Domänen in nicht-ribosomalen Peptidsynthetasen sowie biochemische und strukturelle Untersuchungen zu gespaltenen Inteinen
Volltext
Abbildung
ÄHNLICHE DOKUMENTE
• sind (fast) immer Proteine (Ausnahme: katalytisch aktive RNAs). • haben ein
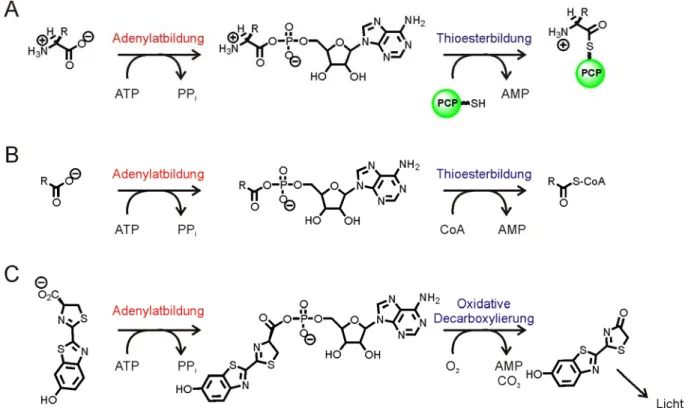
• knüpfen C–C oder C–O oder C–N- Bindungen bei gleichzeitiger Spaltung von ATP (als
Nicht nur bei aku- ten Sportverletzungen wie Zerrungen oder Prellungen ist die hochdosierte Enzymkombination eine gute
Gegenanzeigen: Bei bekannter Über- empfindlichkeit gegen einen der arzneilich wirksamen Bestandteile, gegen Ananas-Früchte oder einen der Hilfsstoffe darf Wobenzym ® plus
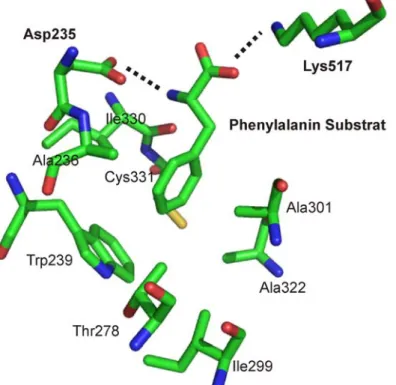
Ist eine dieser kon- servierten Aminosäuren deletiert oder durch eine andere Aminosäuren ausgetauscht, wird der Abbau von β -Catenin verhindert, da β -Catenin nicht mehr an
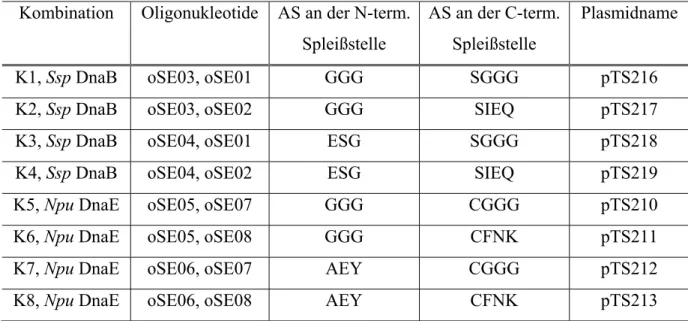
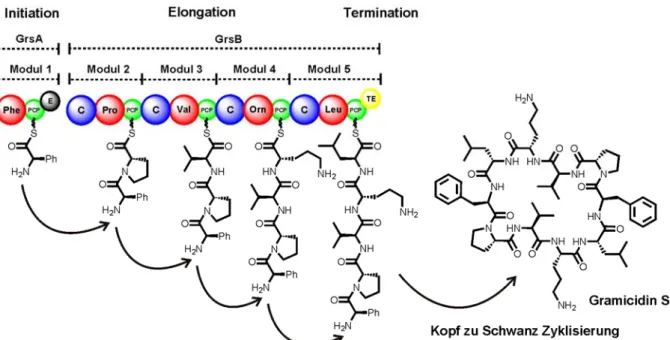
von Bacillus brevis ATCC 8185 als Templat mit den Primern P21 und P5 verwendet. Die Klonierung erfolgte über SphII und BamHI in einen pQE70 Vektor. XIV) p[TycB 3 -ATE]:
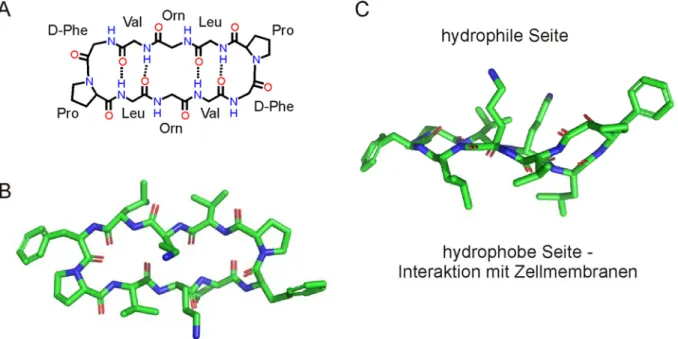
Da das Peptid aufgrund seiner geringen Größe nicht daran gehindert wird, in den Kern zu gelangen, kann bei einer diffusionsunterstützten Verteilung davon ausgegangen werden, daß
Bisher konnte eine Vielzahl an Naturstoffen aus Pilzen isoliert und identifiziert werden, an deren Biosynthese nicht nur NRPS oder PKS/NRPS Enzyme sondern auch
![Abbildung 13 NMR-Strukturen der apo- und holo- TycC3 PCP-Domäne [87]. Sowohl die apo-, als auch die holo-Form befinden sich im langsamen Austausch zwischen zwei Konformationen](https://thumb-eu.123doks.com/thumbv2/1library_info/3676168.1504688/32.892.112.790.410.638/abbildung-strukturen-tycc-domäne-befinden-langsamen-austausch-konformationen.webp)
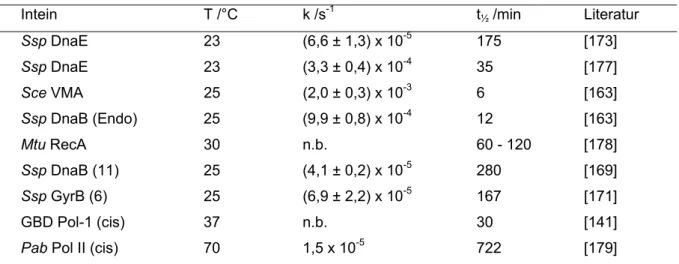
![Abbildung 22 Proteinsemisynthese mit Hilfe künstlich gespaltener Inteine. A) Schematische Darstellung der N-terminalen Modifikation von Proteinen mit dem Ssp DnaB Intein (gespalten an Position 11) [169]; B) Schematische Darstellung der terminalen Modifik](https://thumb-eu.123doks.com/thumbv2/1library_info/3676168.1504688/51.892.154.737.509.778/abbildung-proteinsemisynthese-gespaltener-schematische-darstellung-modifikation-schematische-darstellung.webp)