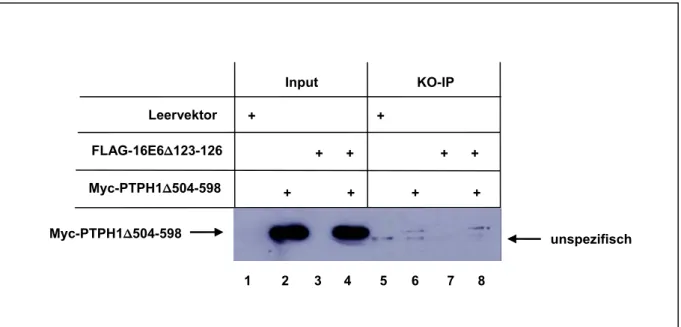
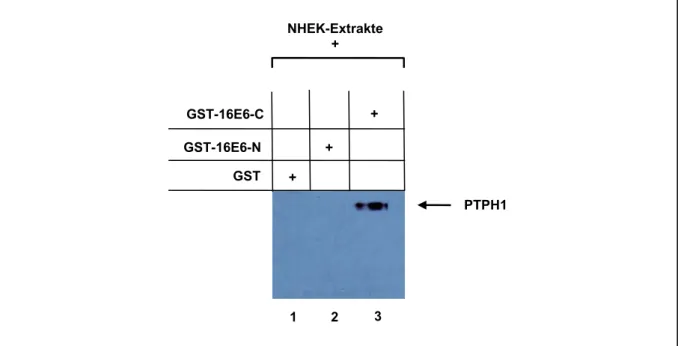
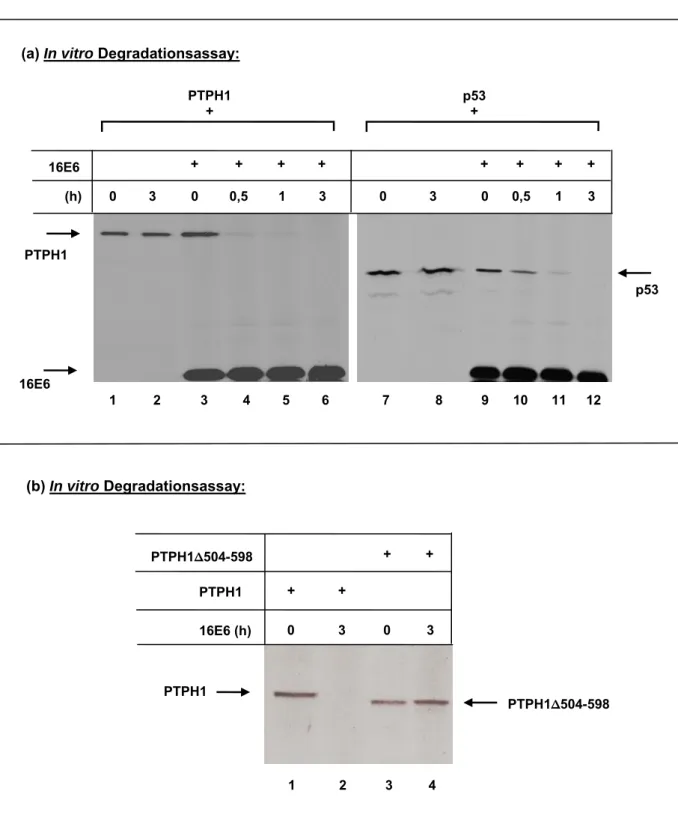
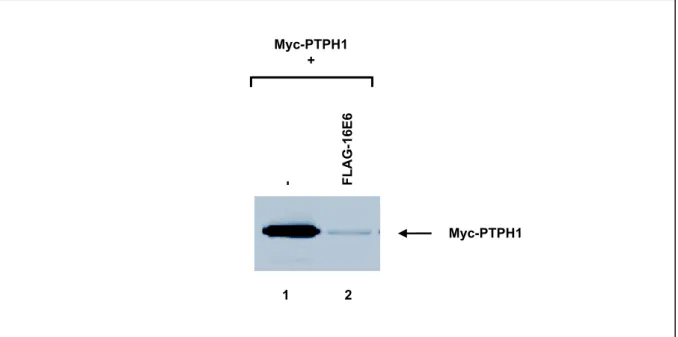
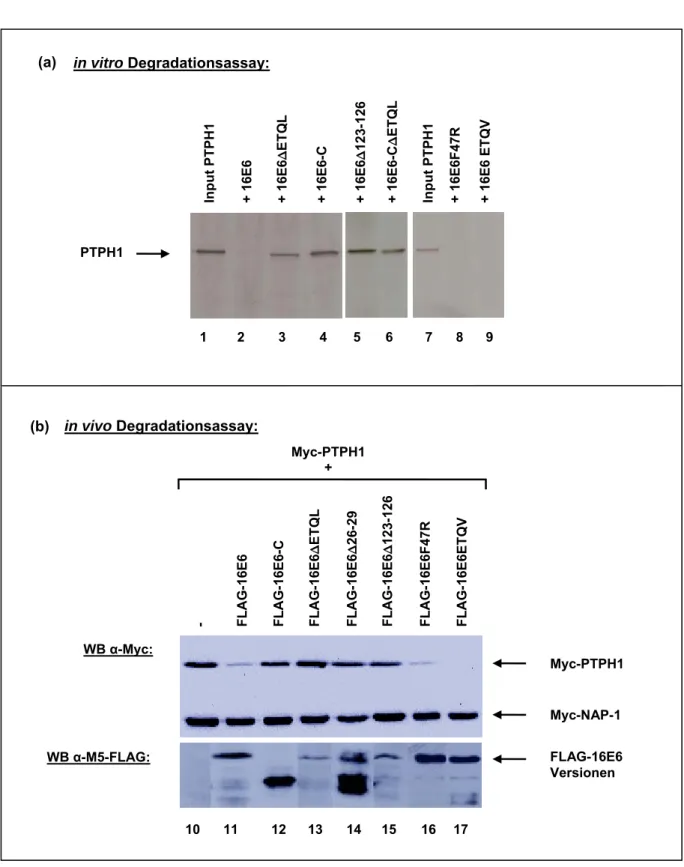
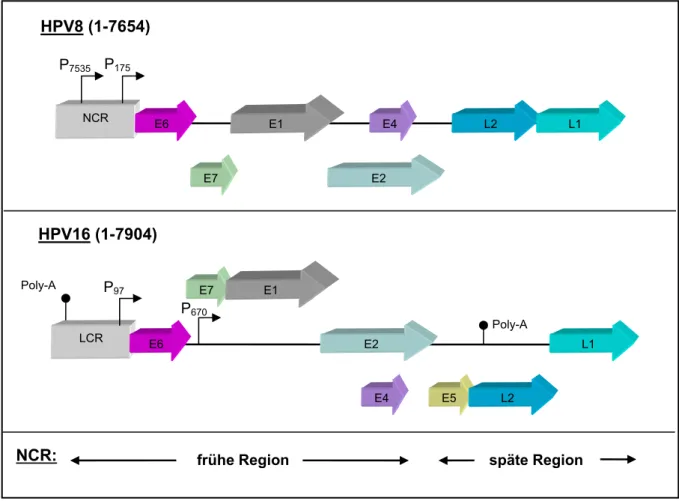
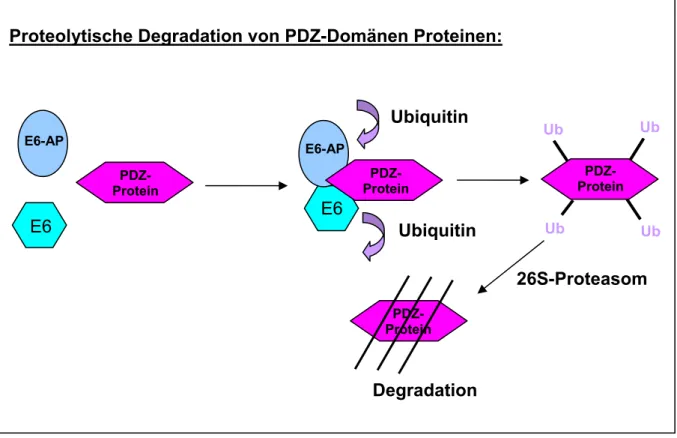
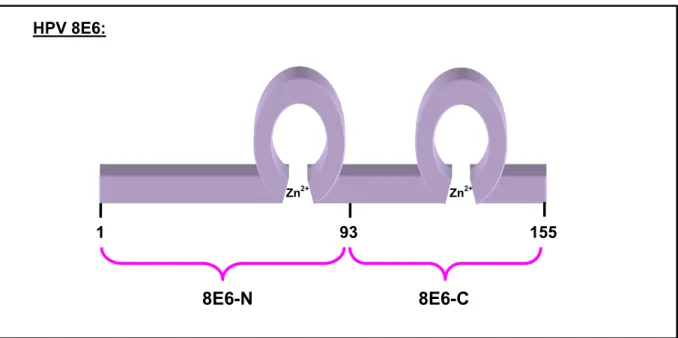
Charakterisierung der Interaktion der E6- Onkoproteine humaner Papillomviren mit der Protein-Tyrosin-Phosphatase H1 (PTPH1)
Volltext
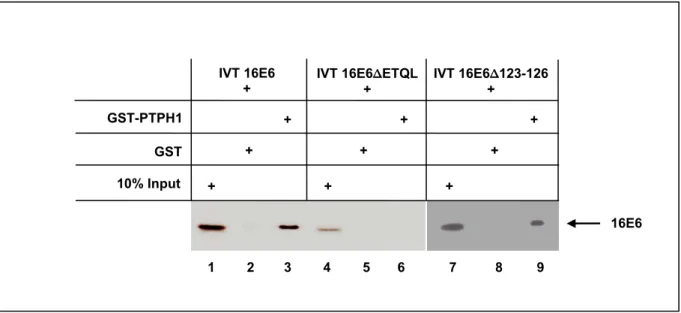
Abbildung
ÄHNLICHE DOKUMENTE
Die umstrittene Er- nennung von 13T Reema Dodin 13T , einer Amerikanerin mit palästinen- sischen Wurzeln, als Teil seines Teams für Gesetzgebungsfragen, deutet neben den Plänen
Gegenanzeigen: FeniHydrocort Creme 0,5 % darf nicht angewendet werden bei bekannter Überempfi ndlichkeit gegen den Wirkstoff Hydrocortison oder einen der sonstigen Bestandteile,
7.1.4 Engagement, Handlungs-, Bearbeitungs- und Problemlösungsstrategien der Lehrer ...
Die quali- tative Studie untersucht auf der Grundlage von ExpertInneninterviews die Theorien, die LehrerInnen bezüglich einer erfolgreichen oder weniger erfolgreichen Gestaltung des
Aber wenn jetzt das vierte, fünfte Kind auf der Welt ist und die Frauen keine Angst mehr haben müssen, dass die, die sie bereits haben, sterben werden, dann wollen sie keine
Die Siegernation ist weit entfernt von jener Regenbogennation, die sich Mandela bei seinem Amtsantritt zum Ziel gesetzt hatte: „Wir gehen die feier- liche Verpflichtung
Als Baschar al-Assad im Jahr 2000 die Nachfolge seines Vaters Hafis al- Assad antrat, hofften viele, dass der junge, in London ausgebildete Augen- arzt die Lösung einiger
„Es ist für uns ein großes Anliegen, dass wir für unsere Beschäftigten und damit für deren Angehörige und unsere Patienten durch eine möglichst