Charakterisierung der Rolle zellulärer Proteine bei der Transkription und Replikation von Papillomviren
Volltext
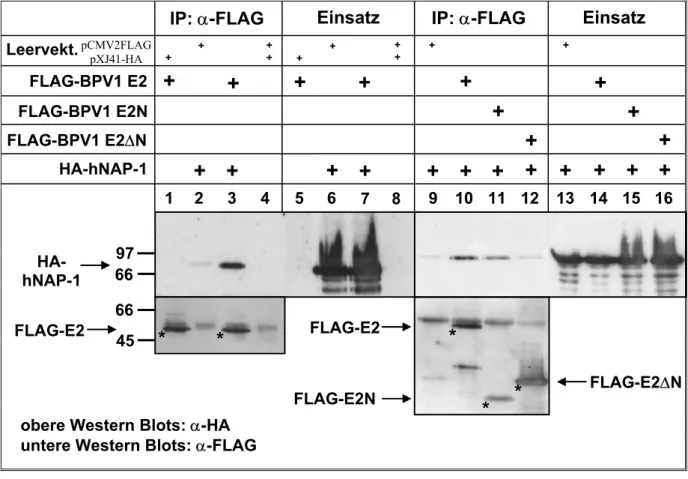
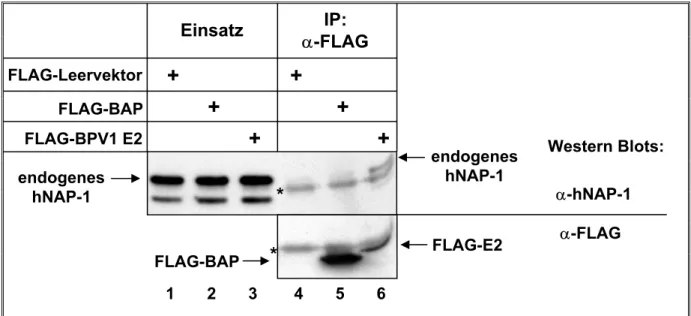
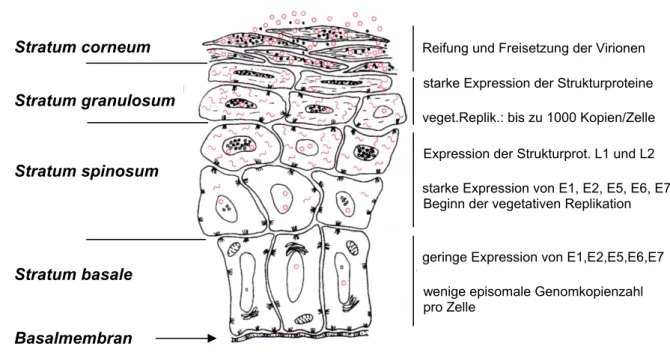
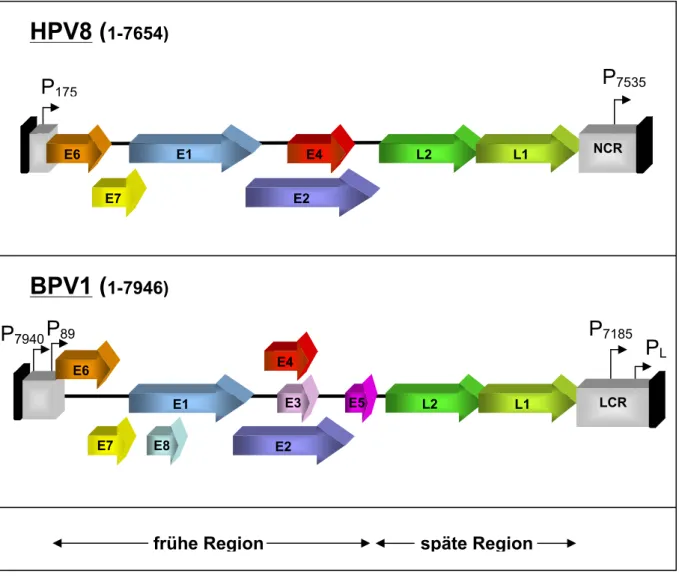
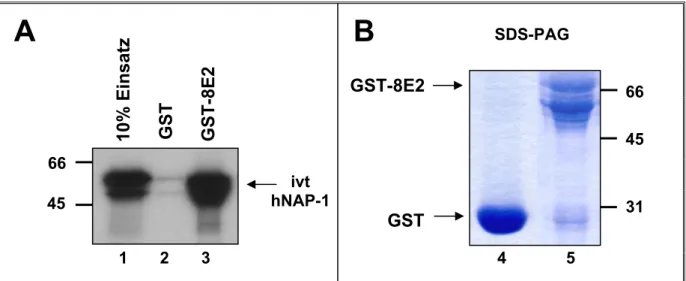
Abbildung

ÄHNLICHE DOKUMENTE
Die Aufrechterhaltung der Proteinhomöostase (Proteoastase) ist von zentraler Bedeutung
Diesbezüglich ergaben sich durch diese Arbeit auch Hinweise darauf, dass die Funktionalität von CHN-1 in verschiedenen Komplexen durch dessen quartäre Struktur
Während für DCs bereits gezeigt wurde, dass sie die Differenzierung zu adaptiven regulatorischen T Zellen (Tregs) induzieren können, konnte den durch B Zellen tolerisierten
Gleichzeitig konnte nachgewiesen werden, dass Zellen durch die verstärkte Palmitoylierung von BAX gegenüber einem Apoptose-Stimulus sensibilisiert wurden und diese mit einer
Zygomycota Incertae sedis Mortierellales Mortierellaceae unidentified Mortierellaceae sp 10060 saprophytic (Jacobs et al. 2010; Vadivelan &. Venkateswaran 2014) Zygomycota
[r]