DNA-bindende Proteinstrukturen DNA-bindende Proteinstrukturen
(und ihre Rolle bei Signaltransduktion und Transkription) (und ihre Rolle bei Signaltransduktion und Transkription)
Ein Vortrag im Rahmen des F1-Praktikums
„Biochemie“ an der Johannes-Gutenberg-Uni-Mainz Vortragender: Christian Lehmann
Betreuer: Claudia Prinzen
Public Documentation License Notice
The contents of this Documentation are subject to the Public Documentation License Version 1.0 (the "License"); you may only use this Documentation if you comply with the terms of this License. A copy of the License can be found at
http://www.openoffice.org/licenses/PDL.html.
The Original Documentation is "DNA-bindende Proteinstrukturen".
The Initial Writer of the Original Documentation is Christian Lehmann Copyright © 2006. All Rights Reserved.
Initial Writer contact(s): LComputer@t-online.de
DNA-bindende Proteinstrukturen
Inhalt des Vortrages 1
Einordnung von Transkriptionsfaktoren Allgemeines, Anforderungen
DNA-bindende Motive der Transkriptionsfaktoren DNA-Erkennung
Helix-Turn-Helix (HTH) / Homöodomänen Zinkfingermotiv
Steroidrezeptoren Leucin-Zipper
Helix-Loop-Helix (HLH) Beispiele zu den Motiven
DNA-bindende Proteinstrukturen
Inhalt des Vortrages 2
Spezielle Regulationsmechanismen
Wirkungen von Transkriptionsfaktoren
Entwicklung von Geweben (Zelldifferenzierung) Reaktionen auf äußere Reize
Aktivierung durch Phosphorylierung Aktivierung durch Ligandenbindung Aktivierung durch Proteolyse
Aktivierung durch Inhibitorentfernung Quellen
Fragen
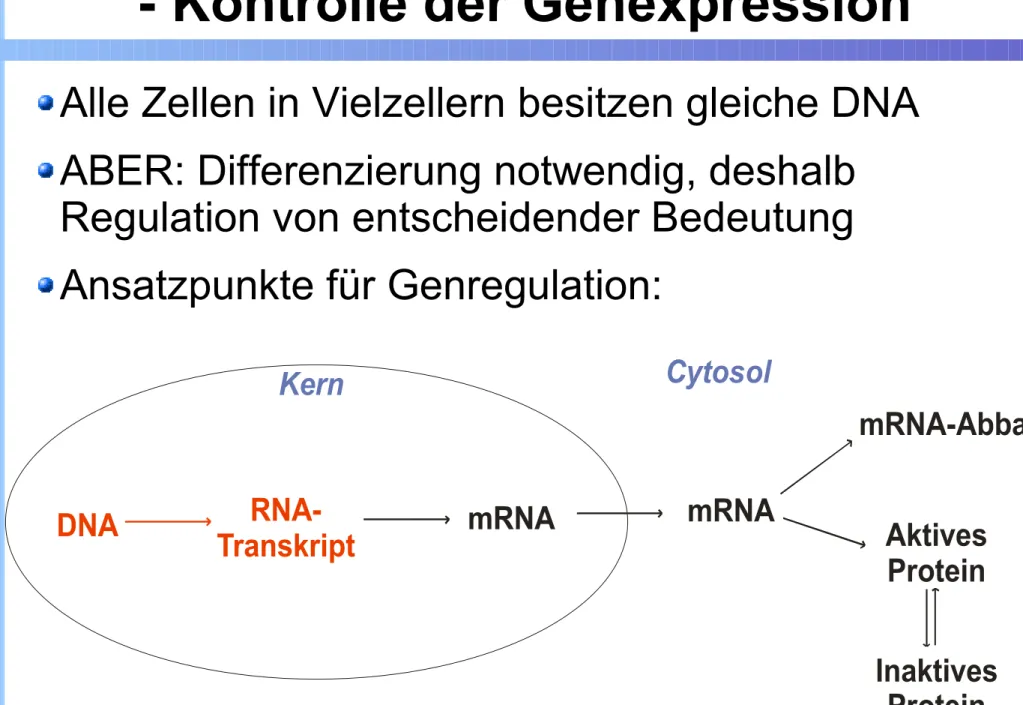
Einordnung von Transkriptionsfaktoren - Kontrolle der Genexpression
Alle Zellen in Vielzellern besitzen gleiche DNA ABER: Differenzierung notwendig, deshalb
Regulation von entscheidender Bedeutung Ansatzpunkte für Genregulation:
DNA RNA-
Transkript mRNA
Kern Cytosol
mRNA
mRNA-Abbau
Aktives Protein
Inaktives Protein
Abbildung verändert nach "Molekulabiologie der Zelle", Alberts, 2004, Köln, Seite 438
Einordnung von Transkriptionsfaktoren – Anforderungen an diese
Was muss ein Transkriptionsfaktor (TF) leisten?
Reaktion auf äußere Einflüsse
(Integration verschiedener Faktoren)
Beeinflussung der Transkription z.B. durch:
Bindung an die DNA
Bindung an andere, vermittelnde Faktoren Flexibilität
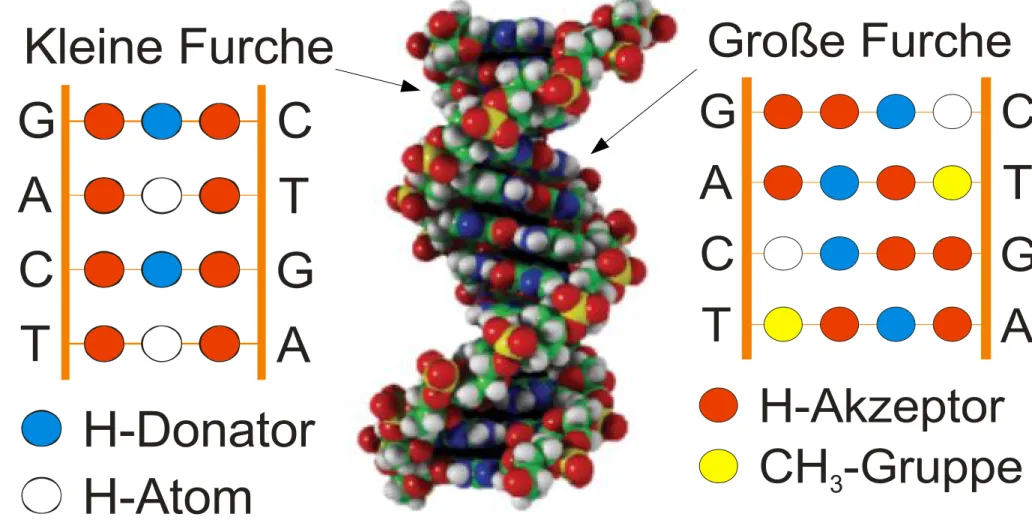
DNA-bindende Motive - DNA-Erkennung
G
G A
C
T A
T C Große Furche
H-Akzeptor CH -Gruppe
3G
G A
C
T A
T C Kleine Furche
H-Donator H-Atom
Abbildung verändert nach "Molekulabiologie der Zelle", Alberts, 2004, Köln, Seite 440
DNA-bindende Motive
- Helix-Turn-Helix-Motiv (HTH)
Als erstes DNA-bindendes Motiv entdeckt
Eines der einfachsten und häufigsten Motive Beinhaltet eine „erkennende“, längere und eine kürzere Helix, über kurzes
Zwischenstück verbunden
Außerhalb des Motivs variable Proteingestalt Teilweise andere Wechselwirkungen mit
DNA
Ermöglicht Feinabstimmung
Oft Bindung als symmetrische Dimere, dadurch vervierfacht sich die Affinität
Abbildung verändert nach "gibk26.bse.kyutech.ac.jp", 25.03.2006, 11:13 Uhr
DNA-bindende Motive - HTH in Homöodomänen
Name nach „Selektorgenen“ bei Drosophila Wichtige „Entwicklungsschalter“
Bei Sequenzierung dieser identisches Stück aus 60 AS gefunden - „Homöodomäne“
Später auch in anderen Organismen gefunden
Spezialfall von HTH, bei der umgebende
Proteinstruktur konserviert ist (Konformation in z.B. Hefe und Drosophila ähnlich, obwohl nur 16 von 60 As identisch)
Abbildung verändert nach "gibk26.bse.kyutech.ac.jp", 25.03.2006, 11:13 Uhr
DNA-bindende Motive - Zink-Finger-Motiv
Zinkionen als Strukturkomponente
Zinkion komplexiert von 2 His und 2 Cys Verbindet meist α-Helix und β-Faltblatt α-Helix bindet in große Furche
Oft im Verbund mit weiteren Zinkfinger Vorteil: Wechselwirkung kann durch Anzahl der Motivwiederholungen
reguliert werden
Keine Dimer-Bildung
Abbildung nach "http://www.chemie.tu-darmstadt.de/akplenio/moproc/zink/zinkfinger/zf_l6.htm", 26.03.2006, 14:10 Uhr
DNA-bindende Motive - Steroidrezeptoren
Ebenfalls Zinkionen in ihrer Struktur, aber komplexiert von 4 Cys
Bildet 2 α-Helixes
Eine α-Helix bindet in große Furche Die andere zuständig für Vermittlung
der Dimerbildung, die bei diesen wieder eine große Rolle spielt
Vorteil: Feinregulation und Variation
durch Dimerpartner möglich – 2. α-Helix (Homodimere vs. Heterodimere)
Abbildung verändert nach "Molekulabiologie der Zelle", Alberts, 2004, Köln, Seite 447
DNA-bindende Motive - Leucin-Zipper
Dimerbildung zur DNA-Erkennung weit verbreitet, oft in verschiedenen Domänen Leucin-Zipper vereint DNA-Erkennung und Dimerisierung in einem Motiv
Verbindung zweier α-Helixes (je eine von jedem Monomer) durch hydrophobe
Seitenketten (meist Leucin)- coiled coil Unterhalb Trennen der Helixes und
Bilden einer Y-Struktur zur DNA-Bindung mit ihren Seitenketten
Abbildungen nach "www.zoologie.uni-bonn.de/Neurobiologie/boehm/online-sem/ieg/sld009.htm", 23.03.06, 10.00 Uhr
DNA-bindende Motive - Helix-Loop-Helix (HLH)
Verwandt mit Leucin-Zipper
Kürzere α-Helix, Schleife, längere α-Helix Längere faltet sich gegen die kürzere
Struktur bindet sowohl an DNA als auch an zweites HLH-Motiv
Sowohl Homo- als auch Heterodimere Längere α-Helixes ausgehend von der Dimerisierungebene stellen spezifische DNA-Kontakte her
Basische HLH in der Lage DNA zu binden
Abbildung nach "http://neuroplasticity.gwdg.de/x3e1ef37e9c552/", 26.03.06, 15:20 Uhr
DNA-bindende Motive - Beispiele 1
SP1 – Zink-Finger:
3 Zink-Finger hintereinander
Allgemeiner Transkriptionsfaktor Bindet an GC-Box
Meist verantwortlich für eine basale Genaktivität Retinsäurerezeptor – Steroidrezeptor:
Verantwortlich für Morphogenese (z.B. beim Huhn)
Erkennt Vitamin-A-Derivat („Vitamin-A-Säure“) Verschiedene Iso-Formen für verschiedene Zielsequenzen
DNA-bindende Motive - Beispiele 2
AP1 – Leucin-Zipper:
Allgemeine Transkriptionsfaktorfamilie (z.B. fos, jun)
Reagiert auf Phorbolester Bindet an TRE-Sequenzen
z.B. verantwortlich für Knochenausbildung SREBP – Helix-Loop-Helix:
Vermittelt Cholesterin-Mangel-Infomation
Sorgt für die Synthese der Enzyme, die für die autonome Cholesterinsynthese verantwortlich zeichnen
Spezielle Regulationsmechanismen
- Wirkungen von Transkriptionsfaktoren
Beruhen fast immer auf der Regulation der Initiation der Transkription der Gene
Basaler Initiationskomplex (RNA-Polymerasen können nicht autark an die DNA binden)
Transkriptionsfaktoren sorgen für Positionierung
Binden an Promotor oder an weitere Sequenzen (z.B.
Enhancer- / Response- oder upstream-Faktoren) Oftmals weite Entfernung, deshalb DNA-Schlaufen Manchmal keine direkte DNA-Bindung, sondern Bindung an andere Faktoren
Auch Chromatinstruktur entscheidend für Transkription
Spezielle Regulationsmechanismen - Entwicklung von Geweben
Transkriptionsfaktoren steuern meist mehrere Gene und ein Gen wird meist durch mehrere TF gesteuert Spezialisierung wird meistens durch einen TF
ausgelöst, der eine Reihe an TFs komplettiert Umwandlung verschiedener Zelltypen nur dann möglich, wenn alle benötigten TFs vorhanden Positive Rückkopplung, DNA-Methylierung und Chromatinstrukturvererbung sind Zellgedächtnis Später werden äußere Signale in verschiedenen Zelltypen unterschiedlich verarbeitet
z.B. der Glucocortikoidrezeptor wirkt in Leber auf andere Gene als in anderen Zellen
Spezielle Regulationsmechanismen - Reaktionen auf äußere Reize
TFs müssen auf Signale von außen reagieren Verschiedene Spielarten bekannt:
Proteinsynthese Ligandenbindung Phosphorylierung
Hinzufügen von Untereinheiten Demaskierung vom Inhibitor
Ermöglichen der Transports in den Kern Freisetzen von einer Membran
Kurze Erläuterung mit einigen kurzen Beispielen
Spezielle Regulationsmechanismen
- Modifizierung durch Phosphorylierung
Phosphorylierung ermöglicht oder verhindert Aktivität dieser Transkriptionsfaktoren
Beispiel: CREB (cAMP response element–binding) Gehört zur Gruppe der Leucinzipper-Proteine
Wird in verschiedene Zellen durch verschiedene Signalkaskaden aktiviert durch Phosphorylierung (z.B. durch RSK2 oder (MAPKAP) Kinase-2)
Bindet an CRE-Kontrollsequenzen der entsprechenden Gene
Wichtige Funktion in frühen Wachstumsprozessen (z.B. bei der Entwicklung von Mäusen)
Spezielle Regulationsmechanismen
- Modifizierung durch Phosphorylierung
Abbildung nach
"www.dddmag.com/PRArchivebyIssue.aspx?RELTYPE=CVS&YEAR=2003&MONTH=10", 27.03.06
Spezielle Regulationsmechanismen - Aktivierung durch Ligandenbindung
Ligandenbindung ermöglicht Funktion des TFs Beispiel: Retinsäurerezeptor - RAR
Bindung des Liganden über elektrostatische Wechselwirkungen, dadurch Umlagerung des Rezeptorproteins (=Deckel zuklappen)
Neue Oberfläche, dadurch Bindung von Co-
aktivatoren, die die eigentliche Wirkung vermitteln Spielt Rolle bei Musterbildung, Differenzierung und dem Wachstum von Zellen
Wesentlich für Reproduktion und Sehvorgang
Retinsäure wird gebildet aus Vitamin A und kann nicht de novo synthetisiert werden
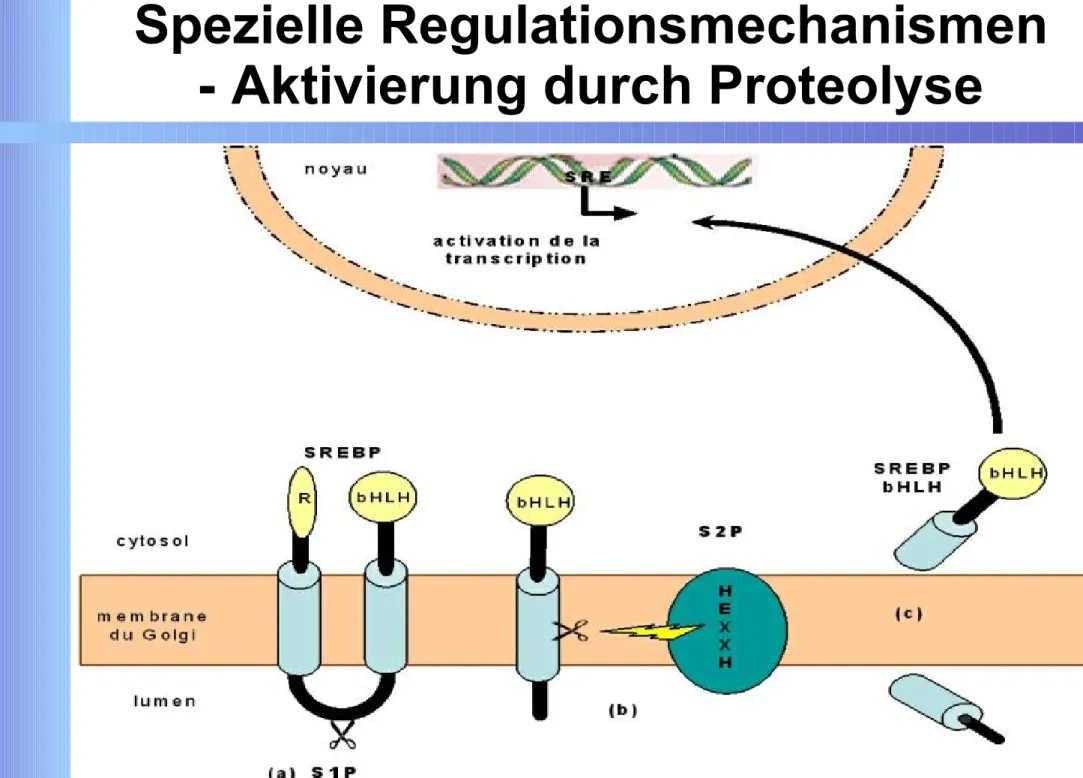
Spezielle Regulationsmechanismen - Aktivierung durch Proteolyse
Proteolytische Spaltung ermöglicht Funktion
Beispiel: SREBP (sterol regulatory binding proteine) Homodimer, lokalisiert in ER-Membran
Herrscht Cholesterinmangel kann SCAP nicht mehr an Cholesterin binden und spaltet mit einer
membrangebundenen Serinprotease (S1P) SREBP Dadurch SREBP frei in ER-Membran beweglich
Kann dadurch eine Zink-Metalle-Protease erreichen Dadurch Abspaltung eines basischen HLH-TF
Bindung an SRE und Aktivierung der Synthese von z.B. HMG-CoA-Reduktase, HMG-CoA-Synthase, Prenyltransferase und des LDL-Rezeptors
Spezielle Regulationsmechanismen - Aktivierung durch Proteolyse
Abbildung nach "http://www.theses.ulaval.ca/2003/21080/ch01.html", 27.03.06, 15.30 Uhr
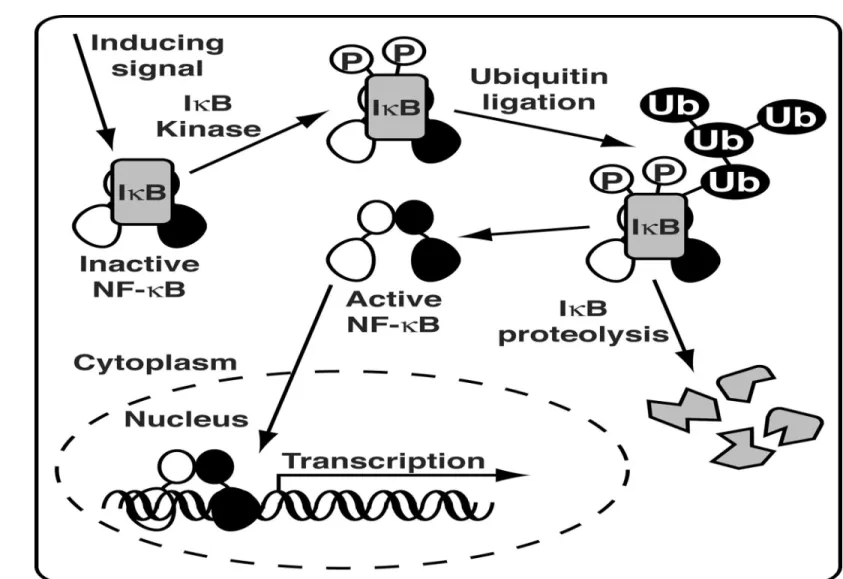
Spezielle Regulationsmechanismen - Aktivierung durch Inhibitorentfernung
Inhibitoren können Funktion oder die Wanderung in der Kern verhindern
Beispiel: NFκB (sterol regulatory binding proteine) Ubiquitär, herausragend bei Immunantworten
Reguliert u.a. Interleukine und Wachstumsfaktoren, Zytokine und Zelladhäsionsrezeptoren, Apoptose
Kerntransportsequenz durch IκB geschützt
Durch Signale von außen Ubiquitinierung von IκB, dadurch Abbau und Freisetzung von NfκB
Wanderung in Kern und Aktivierung der Zielgene
Spezielle Regulationsmechanismen - Aktivierung durch Inhibitorentfernung
Abbildung nach "http://www.chemistry.sdsu.edu/faculty/Huxford/", 27.03.06, 15.40 Uhr
Quellen
Lewin B., „Molekularbiologie der Gene“, Spektrum, 2004 Alberts B., „Molekularbiologie der Zelle“, VCH, 1998
Stryer L., „Biochemie“, Spektrum, 2003
Doktorarbeit Sonnenhauser S., „Analyse der NF-κB Funktion durch
konditionale Geninaktivierung in CCL17+ dendritischen Zellen der Maus“, Technische Universität München, 02.06.2005
Doktorarbeit Kapelle M., „Isolierung und Charakterisierung des humanen Waisen-Kernrezeptors Germ Cell Nuclear Factor (hGCNF)“, FU Berlin, 12.
April 2002 (http://www.diss.fu-berlin.de/2002/143/index.html)
Doktorarbeit Bleckmann S.,“Untersuchungen zur Funktion von cAMP Response Element Binding Protein (CREB) und Activating Transcription Factor 1 (ATF1) während der Embryonalentwicklung der Maus“, Ruprecht- Karls-Universität Heidelberg, Januar 1999
„http://www.ub.uni-heidelberg.de/archiv/1781“, 25.03.2006 Wikipedia
DNA-bindende Proteinstrukturen DNA-bindende Proteinstrukturen
(und ihre Rolle bei Signaltransduktion und Transkription) (und ihre Rolle bei Signaltransduktion und Transkription)
Public Documentation License Notice
The contents of this Documentation are subject to the Public Documentation License Version 1.0 (the "License"); you may only use this Documentation if you comply with the terms of this License. A copy of the License can be found at
http://www.openoffice.org/licenses/PDL.html.
The Original Documentation is "DNA-bindende Proteinstrukturen".
The Initial Writer of the Original Documentation is Christian Lehmann Copyright © 2006. All Rights Reserved.
Initial Writer contact(s): LComputer@t-online.de