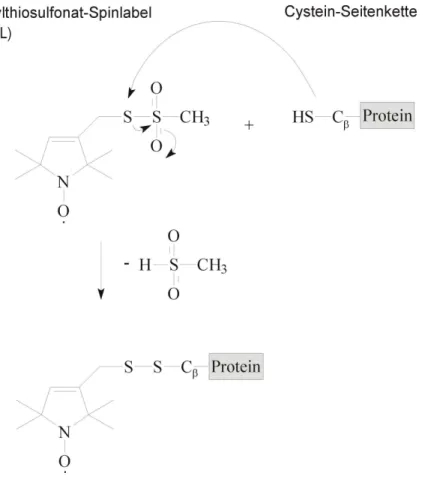
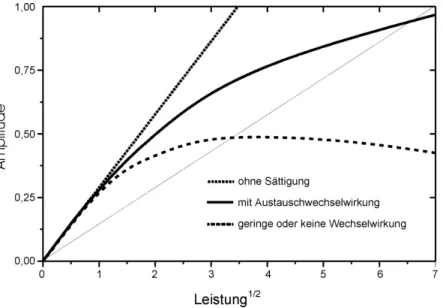
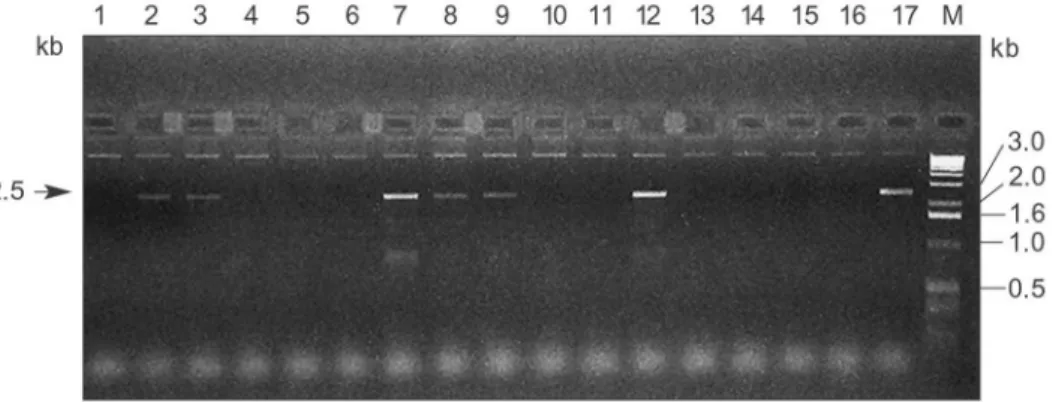
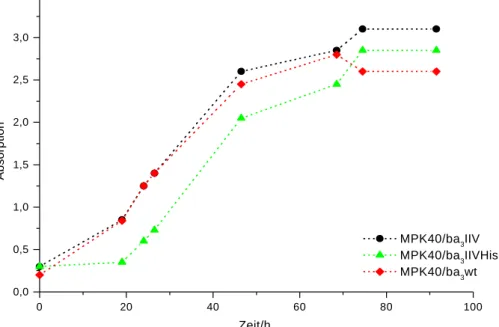
Untersuchungen zur Struktur-Funktionsbeziehung an archaebakteriellen Proteinen der oxidativen Phosphorylierung und Signaltransduktion
Volltext
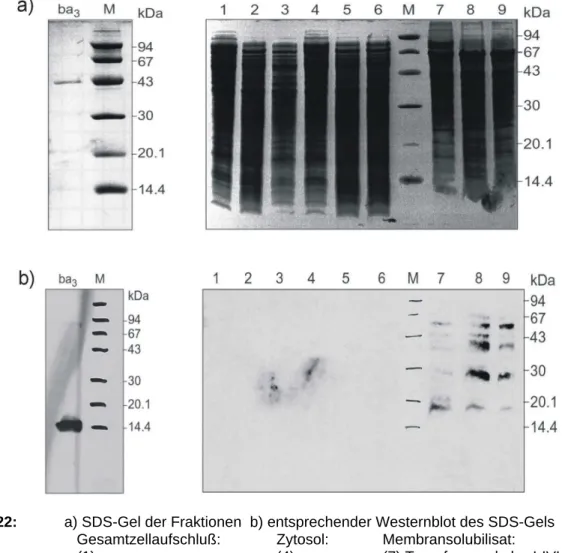
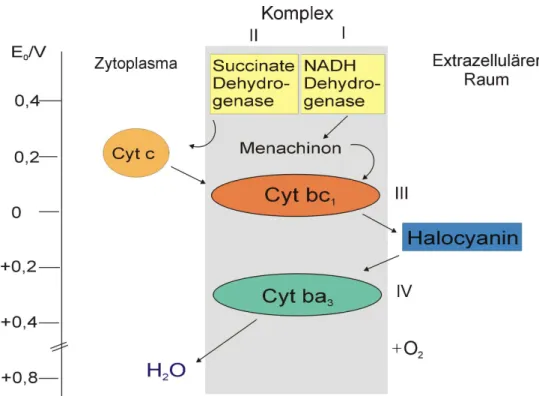
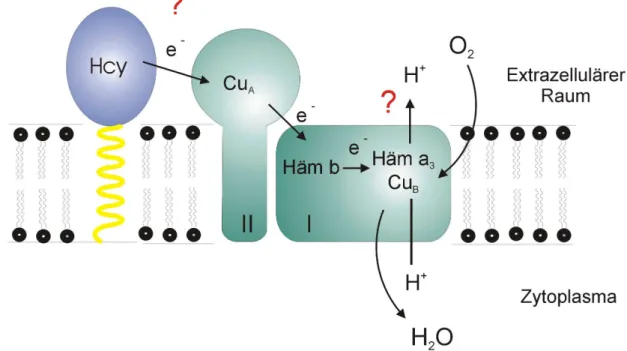
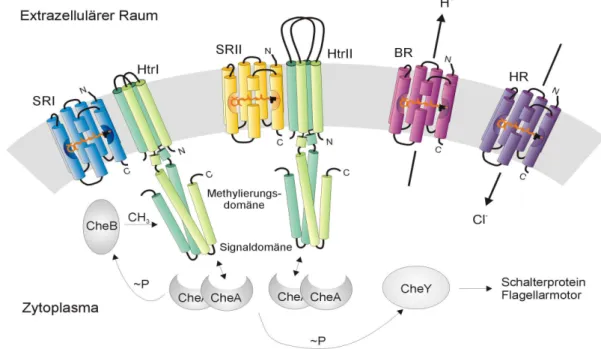
Abbildung
ÄHNLICHE DOKUMENTE
gehabt hat, und wenn von einem guten Chronometer die Rede ist, dem man wirklich einen mittleren, keinen allmählich in einerlei Sinn zunehmenden Aenderungen unterworfenen Gang
Bisher wurde die Funktion des CDeT11-24 Proteins und die Funktion der Phosphorylierung nicht untersucht. Es wird jedoch vermutet, dass das Protein eine ähnliche Funktion
Moleküle (gesteinsartiges Material könnte man nicht essen), Atome (da Metall), Moleküle (keine andere Wahl),
Bei der Messung der verz¨ ogerten Koinzidenzen wird durch das erste Signal (14,4 keV Photon) eine Uhr gestartet, die durch das zweite Signal (122 keV Photon) gestoppt wird. Da
Zur Bestimmung der absoluten (das heißt durch Effekte wie Selbstabsorption und R¨ uckstreuung nicht verf¨ alschten) Aktivit¨ at aus den tats¨ achlich gemessenen Aktivit¨ aten
Information über erhöhte Wasserstände für die gesamte Küste um 70 cm, in der Lübecker Bucht 90 cm über dem mittleren Wasserstand für den 24.12.. Abbildung 8
Ces résultats confirment la pertinence de l‟utilisation des acides gras comme biomarqueurs et sug- gèrent la possibilité d‟utiliser un certain nombre d‟entre eux comme indicateurs
Da sich diese Größen sehr schnell verändern, reicht es nicht aus, sie nur einmal jährlich mit der Polarstern zu messen, sondern sie müssen nahezu kontinuierlich erfasst werden..