Untersuchungen an neuartigen Serin-Threonin-Kinase-Inhibitoren für die In-vivo-Visualisierung von Signaltransduktionswegen des quergestreiften Muskels
Volltext
Abbildung
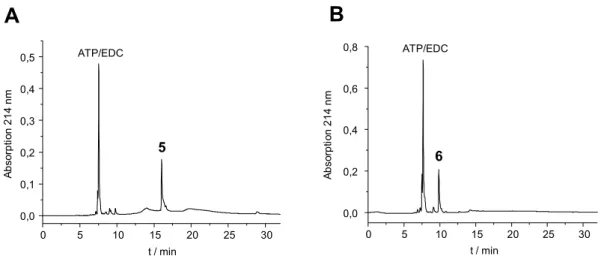
![Abb. 16: HPLC-Chromatogramme und Massenspektren von (A) P 3 -(N ε -(N-FITC-[K5, V6, A7]-CONH 2 - -Kemptide)-lysyl)-P 1 -adenosin-triphosphat 2 und (B) P 3 -(N ε -(N-TRITC-[K5, V6, A7]-CONH 2 -Kemptide)-lysyl)-P 1 -adenosin-triphosphat 3 nach Reinigung](https://thumb-eu.123doks.com/thumbv2/1library_info/3651450.1503322/43.892.137.784.292.828/chromatogramme-massenspektren-kemptide-adenosin-triphosphat-kemptide-triphosphat-reinigung.webp)
![Abb. 17: Autoradiogramme der Verdrängung von [γ- 32 P]-ATP durch ATP und P 3 -(N ε -(N-acyl-O-methyl)-lysyl)- -(N-acyl-O-methyl)-lysyl)-P 1 -adenosin-triphosphat 1 bei der Phosphorylierung von c-C1C2 durch PKA-C](https://thumb-eu.123doks.com/thumbv2/1library_info/3651450.1503322/45.892.275.605.397.713/autoradiogramme-verdrängung-methyl-lysyl-methyl-adenosin-triphosphat-phosphorylierung.webp)
ÄHNLICHE DOKUMENTE
Lavogina D., Lust M., Viil I., König N., Raidaru G., Rogozina J., Enkvist E., Uri A., Bossemeyer D., Structural Analysis of ARC-Type Inhibitor (ARC-1034) Binding to Protein Kinase
Der großen Diversität der antigenspezifischen Rezeptoren und Antikörper des erworbenen Immunsystems liegen genetische Rekombinationsmechanismen
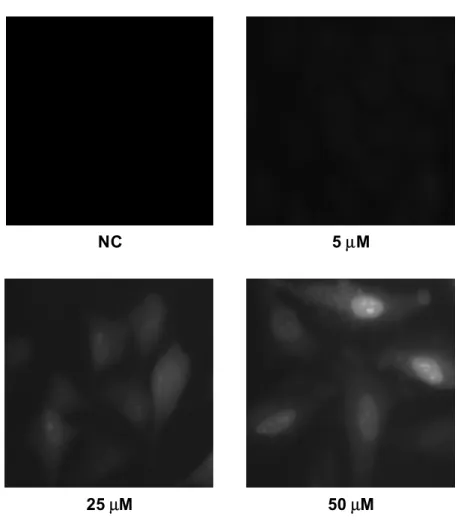
Mögliche Auswirkungen des inflammatorischen Milieus auf Proteinebene in Myotuben wur- den mit immunzytochemischen Nachweismethoden untersucht. Hierzu wurden die pri- mären
Kinetic parameters, characterizing interaction of the bifunctional inhibitor AdoC(Ahx)Arg 6 with protein kinase A in the presence of two substrates, ATP and Kemptide,
ROS are thought to promote atherosclerosis through a variety of mechanisms, including enhanced oxidation of lipoproteins (Steinberg 1997), activation of proinflammatory genes (Marui,
Als alleinige Methode für die Aufreinigung von binären oder multimeren Protein-Protein Komplexen ist die Co-Immunpräzipitation eher ungeeignet, da zum einen hochaffine
Mittels Bindung am Eisen der Hämgruppe und nachfolgender Oxidation zu Fe 3+ wird die Bindung von NO an das Eisen der Hämgruppe und die Aktiverung der löslichen
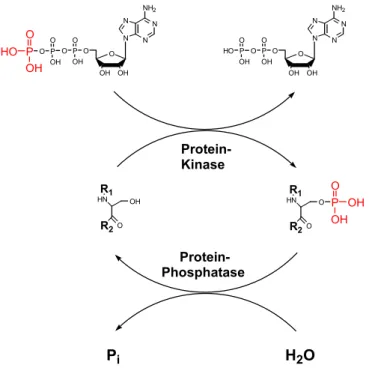
Des Weiteren konnten für diese Aminosäuren sowohl eine Auto-Phosphorylierung als auch eine Phosphorylierung durch eine andere Kinase gezeigt werden, die bei besserer Auflösung
![Abb. 18: Autoradiogramme der Verdrängung von [γ- 32 P]-ATP durch [V6, A7]-Kemptide und N-Fmoc-[K5, V6, A7]-CONH 2 -Kemptide 4 bei der Phosphorylierung von c-C1C2 durch PKA-C](https://thumb-eu.123doks.com/thumbv2/1library_info/3651450.1503322/46.892.247.635.193.526/abb-autoradiogramme-verdrängung-kemptide-fmoc-conh-kemptide-phosphorylierung.webp)
![Abb. 19: Autoradiogramme der Verdrängung von [γ- 32 P]-ATP durch ATP und P 3 -(N ε -(N-FITC-[K5, V6, A7]- A7]-CONH 2 -Kemptide)-lysyl)-P 1 -adenosin-triphosphat 2 bei der Phosphorylierung von c-C1C2 durch PKA-C](https://thumb-eu.123doks.com/thumbv2/1library_info/3651450.1503322/47.892.252.622.405.698/autoradiogramme-verdrängung-fitc-conh-kemptide-adenosin-triphosphat-phosphorylierung.webp)
![Abb. 20: Autoradiogramme der Verdrängung von [γ- 32 P]-ATP durch ATP und P 3 -(N ε -(N-TRITC-[K5, V6, A7]- A7]-CONH 2 -Kemptide)-lysyl)-P 1 -adenosin-triphosphat 3 bei der Phosphorylierung von c-C1C2 durch PKA-C](https://thumb-eu.123doks.com/thumbv2/1library_info/3651450.1503322/48.892.206.663.135.454/autoradiogramme-verdrängung-tritc-conh-kemptide-adenosin-triphosphat-phosphorylierung.webp)
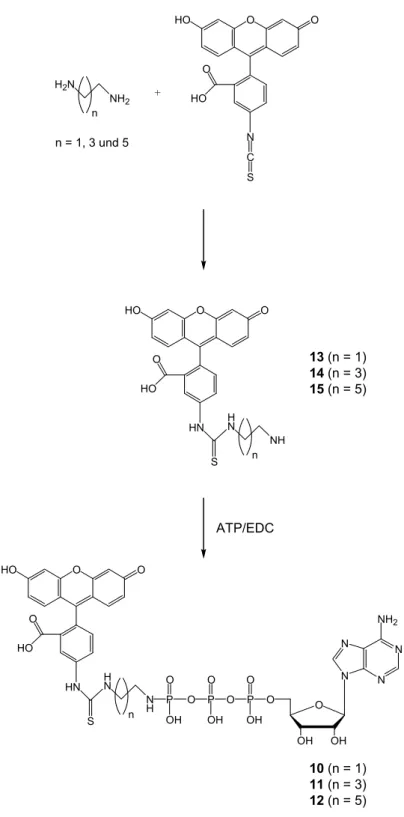
![Abb. 27: Autoradiogramme der Verdrängung von [γ- 32 P]-ATP durch ATP und P 3 -(4-(N-FITC-Aminoethyl)- -(4-(N-FITC-Aminoethyl)-phenylamino)-P 1 -adenosin-triphosphat 19 bei der Phosphorylierung von c-C1C2 durch PKA-C](https://thumb-eu.123doks.com/thumbv2/1library_info/3651450.1503322/56.892.217.660.693.1042/autoradiogramme-verdrängung-aminoethyl-aminoethyl-phenylamino-adenosin-triphosphat-phosphorylierung.webp)