Transcriptional control of the H‐NS antagonists LeuO and RcsB‐BglJ in Escherichia coli
Volltext
Abbildung

ÄHNLICHE DOKUMENTE
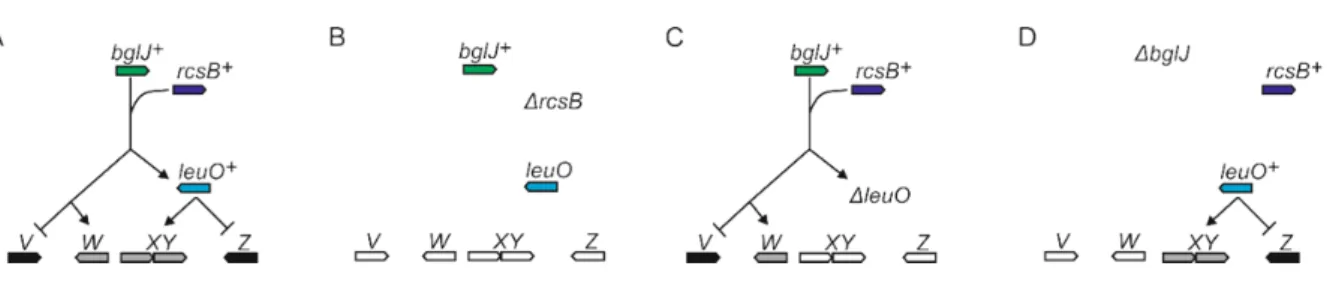
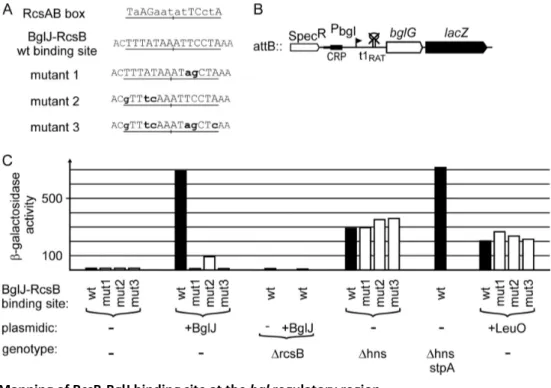
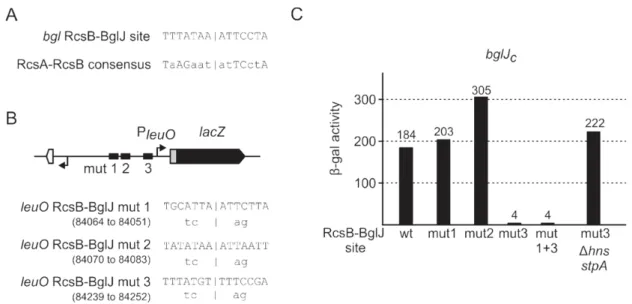
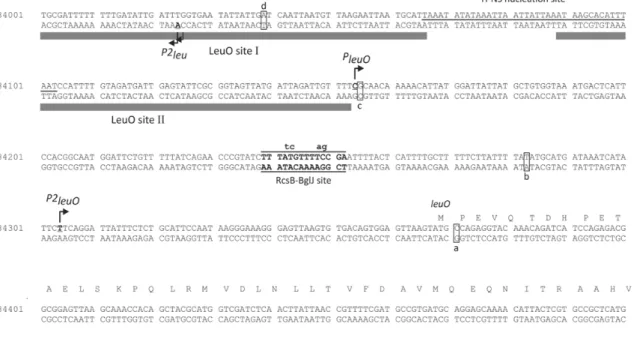
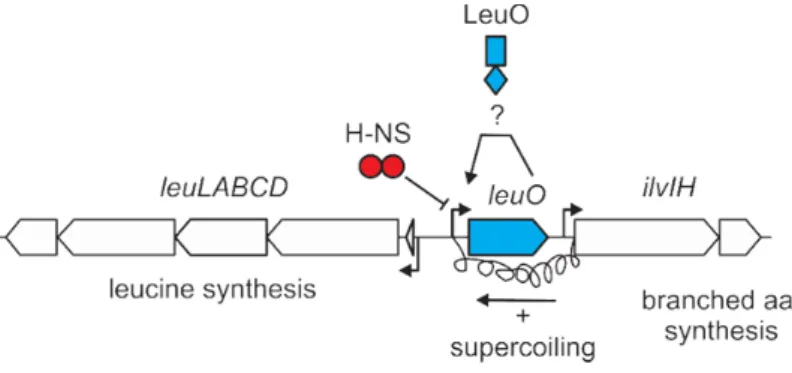
Previous data suggest, that leuO regulation is tightly controlled and involves a double-positive feedback loop, since the leuO gene is activated by BglJ-RcsB,
An increase in the promoter activity with the increase in osmolarity in case of proU, and by the action of specific transcription factors in bgl leading to an increase in
• Simulation of the phase equilibria: Carrying out GEMC-NPT and NVT sim- ulations using the four developed ab initio 5-site intermolecular potentials of hydrogen and fluorine for
One cyano group of the counter anion completes the coordina- tion environment of the caesium atom (Cs-N 3.420(8) ˚ A), while the second one is involved in hydrogen bonding. *
Auf Proteinebene konnten wir nachweisen, dass der C-terminale Knock down von Nesprin-2 zu einer verminderten Expression von Histon H1.2 in HaCaT Zellen
Synthetic promoter sequences did not show as high variation in their expression levels across cells and sites for specific transcription factors were not predicted. To evolve
Large SIMs occur infrequently in randomized networks (Table 1, P < 0.01), because there is a low probability that a large number of operons controlled by a single